same-sense codons: codones
sinónimos.
![]() synonymous
codon.
synonymous
codon.
Sanger method: método de Sanger.
→ ENZYMATIC
SEQUENCING METHOD
satellite DNA : ADN satélite.
ADN del genoma eucariota sin función conocida, formado por unidades
repetidas en serie —no hay consenso en cuanto a la longitud de estas
unidades; según algunas fuentes varían de 5 a 200 pares
de bases— y pueden llegar a ocupar un espacio de hasta cientos de
miles de pares de bases e incluso mayor, lo que otorga a este ADN propiedades únicas,
por ejemplo, la de poder identificarlo como una fracción separada
de la banda principal de ADN en un gradiente de densidad en cloruro de
cesio, de allí la denominación de «satélite» (no
obstante, en los seres humanos, no todas estas secuencias se distinguen
como una banda separada en un gradiente de densidad, tal es el caso del
ADN satélite alfa y del ADN alfoide, que constituye el grueso
de la heterocromatina centromérica en todos los cromosomas humanos).
Representa más del 10 % del genoma eucariota. Se ubica sobre todo
en los centrómeros y los telómeros de los cromosomas. Véanse
highly repetitive DNA, microsatellite y minisatellite.
satellite RNA : ARN satélite.
Pequeña molécula de ARN (aunque de tamaño superior
a 350 nt) que en las plantas vasculares se encapsida con otros virus;
también se conoce con el nombre de «virusoide».
satellite virus : virus
satélite.
Virus defectuoso que necesita de otro virus (por lo general del mismo
género) para poder multiplicarse y encapsidarse.
scRNA : ARNcp.
![]() small
cytoplasmic RNA.
small
cytoplasmic RNA.
scRNP : RNPcp.
![]() small cytoplasmic
ribonucleoprotein
small cytoplasmic
ribonucleoprotein
scyrp : scirp.
Voz coloquial derivada del acrónimo inglés scRNP (small
cytoplasmic ribonucleoprotein).
Sec61: Sec61.
Complejo constituido por tres clases de polipéptidos transmembranarios
(Sec61p, Sec61ß y Sec61g) que forman
el canal del traslocón propiamente dicho. Cuando la secuencia
señal ingresa en el traslocón, el ribosoma se une firmemente
a Sec61, de modo que el poro no queda expuesto al citoplasma. Véase
translocon.
second site mutation: mutación
supresora intragénica.
![]() supressor
mutation.
supressor
mutation.
sequence motif: motivo secuencial.
![]() motif.
motif.
self-splicing
: autoempalme,
autoayuste.
Empalme o ayuste en el que el propio ARN actúa de catalizador
y, por consiguiente, no requiere la actividad de enzimas proteicas. Véase
ribozyme.
selfish DNA : ADN redundante.
![]() junk
DNA.
junk
DNA.
sense codon: codón
codificante.
Codón que codifica un aminoácido. Véase nonsense
codon.
sense RNA: ARN mensajero,
ARN efector.
![]() messenger
RNA (mRNA).
messenger
RNA (mRNA).
Observación: el término sense RNA se aplica
por lo general a moléculas de ARNm. Véase antisense
rna,
antisense-rna control y messenger
RNA (mRNA).
sense strand: cadena codificante.
![]() coding
strand.
coding
strand.
sequenator: secuenciador.
→ SEQUENCER
sequence: secuencia.
Orden de unión de los monómeros en un biopolímero, por
ejemplo, el orden de aminoácidos en un polipéptido (del extremo
N al extremo C) o de nucleótidos en una hebra de ácido nucleico
(del extremo 3’ al extremo 5’).
sequence-tagged sites: sitios
de secuencia identificada.
Segmentos de ADN breves, de unos 200 o 500 bp, compuestos de
una secuencia nucleotídica única, es decir, de una secuencia
que no se repite en todo el genoma.
Observación: desde el año 1987 la PCR se convirtió en
la herramienta indispensable de todo laboratorio de biología
molecular. Esto llevó a Maynard Olson y cols., del National
Research Council (NRC) Committee on the Mapping and Sequencing of the
Human Genome, a sugerir dos años más tarde, en un artículo
publicado en la revista Science, un lenguaje común para
construir mapas físicos a partir de los fragmentos de ADN clonado,
obtenidos por los métodos diversos que a la sazón se
utilizaban para construir mapas físicos de los genomas (contígs,
fragmentos con sitios de restricción inusuales, sondas para
detectar polimorfismos en el ADN o secuencias que hibridaban in situ
con bandas citogenéticas de los cromosomas). El lenguaje común
eran los STS. La idea era hallar dentro de un fragmento de ADN clonado
una secuencia de nucleótidos que lo caracterizara y que no estuviera
repetida en el genoma. La construcción de un mapa físico
se limitaba entonces a determinar el orden y espaciamiento de los segmentos
de ADN identificados de forma unívoca por esa secuencia. Con
este método era virtualmente posible reconstruir el STS en cualquier
momento mediante una reacción en cadena de la polimerasa (PCR)
sobre el ADN correspondiente, con la ayuda de cebadores específicos
de un veintenar de nucleótidos de largo, sin necesidad de disponer
del clon original. Lo anterior resolvía el problema de la conservación
de genotecas voluminosas y del envejecimiento de los clones (clone
obsolescence) y facilitaba la armonización de los datos
de laboratorios que trabajaban con genotecas diversas. Asimismo, era
posible someter cualquier muestra de ADN a una PCR con los mismos cebadores
específicos para ver si disponía de dicha secuencia única
y utilizarla luego como marcador para construir su mapa físico.
Los sitios de secuencia identificada o STS se habían transformado
en los marcadores convencionales del mapa físico del genoma
humano en el momento en que se descubrieron las EST (expressed sequence
tags). En castellano circulan asimismo las variantes «sitios
de secuencia rotulada» y «sitios de secuencia etiquetada».
Véase expressed sequence tags.
sequencer: secuenciador.
Aparato
que sirve para determinar de forma automática la secuencia de
los monómeros que componen un polímero lineal. Existen secuenciadores
automáticos de ADN y de proteínas.
sequencing: secuenciación.
Procedimiento
analítico que permite determinar la secuencia de aminoácidos
de un polipéptido o la secuencia de nucleótidos de una hebra de
ADN o de ARN. Véanse ENZYMATIC SEQUENCING METHOD, CHEMICAL
SEQUENCING METHOD y SOLID-PHASE PEPTIDE SEQUENCING.
sequencing gel: gel de secuenciación.
Gel
de poliacrilamida en el que se resuelven por electroforesis en condiciones desnaturalizantes
los polinucleótidos de distinto tamaño procedentes
de la secuenciación de un ADN.
short hairpin RNA (shRNA) : ARN
horquillado corto (ARNhc).
Molécula de ARN monocatenario que adopta la forma de una horquilla
debido a apareamientos intracatenarios:

Es sustrato de la endorribonucleasa Dícer, que al escindirlo libera
un ARN monocatenario de unos 22 nt denominado «ARN temporal pequeño» (segmento
negro de la figura, el ARNtp). El ARNhc se conoce asimismo con el nombre
de stRNA precursor (pre-stRNA). Véase Dicer, small
temporal RNA y stRNA precursor.
short tandem repeats: repeticiones
cortas en tándem.
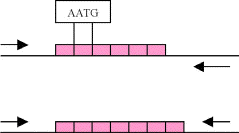 Secuencias
de nucleótidos de dos a cinco pares de bases de longitud y de
función desconocida (AATG en la figura, identificadas como pequeños
rectángulos de color violeta), dispuestas en tándem,
generalmente presentes en regiones no codificantes del genoma. Reciben
asimismo el nombre de «microsatélites». Como el
número de repeticiones varía entre individuos, las regiones
que las contienen (rectángulos violetas de mayor tamaño
en la figura) también son variables, y por eso mismo se llaman «polimórficas»;
en cambio, las regiones flanqueantes son constantes. Los cebadores
de la PCR (flechas) se unen a estas últimas regiones. Véase
DNA profiling.
Secuencias
de nucleótidos de dos a cinco pares de bases de longitud y de
función desconocida (AATG en la figura, identificadas como pequeños
rectángulos de color violeta), dispuestas en tándem,
generalmente presentes en regiones no codificantes del genoma. Reciben
asimismo el nombre de «microsatélites». Como el
número de repeticiones varía entre individuos, las regiones
que las contienen (rectángulos violetas de mayor tamaño
en la figura) también son variables, y por eso mismo se llaman «polimórficas»;
en cambio, las regiones flanqueantes son constantes. Los cebadores
de la PCR (flechas) se unen a estas últimas regiones. Véase
DNA profiling.
shRNA : ARNhc.
![]() short
hairpin RNA.
short
hairpin RNA.
shuttle plasmid: plásmido
versátil, plásmido lanzadera.
Plásmido mixto que funciona como vector de clonación
en células de origen diverso.
Observación: ninguna acepción de la voz «lanzadera» en
el DRAE justifica su traducción por «plásmido lanzadera»,
ni siquiera la sexta: «Medio de transporte rápido, de
ida y vuelta y periodicidad frecuente, entre dos ciudades», pero
es una denominación relativamente frecuente en los libros de
texto especializados. Véase hybrid plasmid.
shuttle vector: vector versátil,
vector lanzadera.
![]() shuttle
plasmid.
shuttle
plasmid.
signal peptide : péptido
señal.
![]() leader
sequence.
leader
sequence.
signal recognition particle: partícula
de reconocimiento de la señal.
Complejo ribonucleoproteico (SRP) del traslocón, que reconoce
la secuencia señal de una proteína de exportación
en vías de síntesis y se une a un receptor ubicado en la
membrana del retículo endoplásmico (receptor de SRP), arrastrando
al ribosoma hacia la membrana del retículo. Consta de seis proteínas
(11S) y de una pequeña molécula de ARN (7S), indispensable
para el ensamblado del complejo. Véase signal
sequence, translocon.
signal sequence : secuencia
señal.
![]() leader
sequence.
leader
sequence.
signature: distintivo.
1. (sust.).
Aminoácido conservado o secuencia de aminoácidos
(motivo proteico) que caracteriza o sirve para reconocer una
proteína o una familia de proteínas. Por ejemplo, se
dice que el aminoácido conservado Lys-220 del motivo D
de las polimerasas dependientes de ARN es el distintivo o la signature de
esas proteínas, o que el motivo B es el distintivo o
la signature de los reguladores ARR que participan en los sistemas
de traducción de señales. Véase motif.
2. (adj.). Que distingue o caracteriza algo. Por ejemplo, el
motivo secuencial único, LSGGQ, característico de los
dominios con actividad ATPasa de los transportadores ABC, recibe en
inglés la denominación de signature sequence o canonical
signature motif de estos transportadores.
Observación: en la primera acepción, no es incorrecta
su traducción literal por «signatura», dado que el
DRAE recoge como primer significado de esta última voz el de «marca
o nota puesta en una cosa para distinguirla de otras». También
se ha propuesto «rúbrica» (en su acepción de «rasgo» o «peculiaridad»).
En la segunda, en cambio, el traductor tiene dos opciones, optar bien
por el calco semántico «signatura» (con el significado
de «distintivo») y entonces se utiliza de forma apuesta: «motivo
signatura» (signature motif), «secuencia signatura» (sequence
motif) o bien traducirlo por el adjetivo «distintivo».
Esta última palabra tiene la ventaja de que también puede
usarse en función sustantiva con el significado de «marca
o señal característica» y quizás transmita
mejor este significado que «signatura».Véanse signature
motif y signature sequence.
signature motif: motivo distintivo.
Observación: se trata de un motivo de aminoácidos
en su 3ª acepción, que define o caracteriza a una proteína
o a un grupo de proteínas, por ejemplo, el motivo distintivo LXXLL
del receptor p160 de hormonas esteroideas (signature motif, LXXLL,
within steroid hormone receptor p160), o el motivo distintivo LSGGQ
de la familia ABC de transportadores transmembranarios (the ABC family
is defined in part by the canonical signature motif LSGGQ whose exact
function remains controversial). Véase motif.
signature sequence: secuencia
distintiva.
Inserción o deleción en la región codificante de
un gen dado que se constata en especies filogenéticamente distantes
y por eso se cree que se ha conservado durante la evolución. Por
consiguiente, puede servir para rastrear el parentesco entre especies
distintas. Véase motif.
silencing trigger : desencadenante
del silenciamiento.
![]() trigger,
RNA interference.
trigger,
RNA interference.
single-stranded DNA (ssDNA) : ADN
monocatenario
Molécula de ADN formada por una sola hebra de desoxirribonucleótidos.
Observación: la sigla española, ADNmc, apenas se
utiliza.
single stranded DNA binding protein: proteína
de unión a ADN monocatenario.
Cada una de las proteínas que se unen a las hebras de ADN recientemente
separadas por la helicasa durante la replicación del ADN. Se colocan
una detrás de otra a lo largo de la hebra separada y forman una
cubierta proteica que mantiene el ADN en el estado de elongación
necesario para que pueda servir de plantilla durante la síntesis
de ADN y de los ARN cebadores.
Observación: en los libros de texto figura como «proteína(s)
SSB», según la denominación inglesa. Véase
DNA replication.
single-stranded RNA (ssRNA)
: ARN
monocatenario
Molécula de ARN formada por una sola hebra de ribonucleótidos.
La mayoría de los ARN son de hebra única.
Observación: la sigla española, ARNmc, apenas
se utiliza.
siRNA : ARNip.
![]() small
interfering RNA.
small
interfering RNA.
siRNP :RNPip.
![]() pre-RISC.
pre-RISC.
Slicer: Eslícer.
Enzima con actividad endorribonucleasa del complejo ribonucleoproteico
RISC. Véase RISC, RNA interference.
Observación: el nombre de esta enzima proviene de un
juego de palabras entre los verbos to dice (cortar en cubitos)
y to slice (cortar en rebanadas).
sliding clamp: abrazadera
deslizante de la ADN polimerasa.
![]() DNA polymerase sliding clamp
DNA polymerase sliding clamp
sliding DNA clamp: abrazadera
deslizante de la ADN polimerasa.
![]() DNA polymerase sliding clamp
DNA polymerase sliding clamp
small cytoplasmic ribonucleoprotein (scRNP,
scyrp) : ribonucleoproteína citoplasmática
pequeña (RNPcp).
Complejo formado por un ARN citoplasmático pequeño (ARNcp)
y proteína(s). Se localiza en el citoplasma de las células
eucariotas. Véase small cytoplasmic RNA.
small cytoplasmic RNA (scRNA) : ARN
citoplasmático pequeño (ARNcp).
Cualquier molécula minúscula de ARN (100 a 300 nucleótidos)
que forma parte de una ribonucleoproteína citoplasmática
pequeña (small cytoplasmic ribonucleoprotein). El único
ARN citoplasmático pequeño identificado hasta la fecha
es el ARNnp 7SL. Este ARNcp forma parte del complejo ribonucleoproteínico
SRP que reconoce los péptidos señal en el retículo
endoplásmico. Véase leader sequence.
small interfering ribonucleoprotein (siRNP)
:ribonucleoproteína interferente pequeña
(RNPip).
![]() pre-RISC.
pre-RISC.
small interfering RNA (siRNA): ARN
interferente pequeño (ARNip).
Pequeños ARNbc de 21 a 25 nucleótidos, resultado de la
fragmentación de un ARNbc de mayor tamaño por parte de
la endorribonucleasa Dicer en el fenómeno de ribointerferencia.
Los dos últimos nucleótidos de cada extremo 3’ quedan
sin aparear —son nucleótidos protuberantes (overhang)— y
sus extremos 5’ están fosforilados. Véase dicer, RNA
interference, small temporal RNA.
small nuclear ribonucleoprotein (snRNP,
snurp) : ribonucleoproteína nuclear pequeña
(RNPnp).
Complejo formado por unas 10 proteínas y una pequeña molécula
de ARN (ARNnp) –que es la que da nombre al conjunto–, presente
en los núcleos de las células eucariotas.
small nuclear RNA (snRNA) : ARN
nuclear pequeño (ARNnp).
Cualquier molécula pequeña de RNA (de 100 a 300 nucleótidos
en los organismos eucariotas superiores y hasta 1000 nucleótidos
en las levaduras) que se localiza en el núcleo de las células
eucariotas. Son indispensables para los procesos de maduración
del ARN, principalmente para el empalme o ayuste (splicing) y la poliadenilación.
small nucleolar RNA (snoRNA) : ARN
nucleolar pequeño (ARNnop).
Pequeña molécula de ARN (de 100 a 300 nucleótidos)
localizada en el nucléolo de una célula eucariota y cuya
presencia es indispensable para el procesamiento de los transcritos primarios
de los ARNr.
small temporal RNA (stRNA) : ARN
temporal pequeño (ARNtp).
Pequeñas moléculas de ARN monocatenario (de 21 a 25 nucleótidos)
que no se traducen en proteína y que desempeñan una función
reguladora al reprimir la traducción de ARNm específicos
en determinados momentos del desarrollo de un organismo. Actúan
bloqueando la traducción del ARNm al unirse con secuencias parcialmente
complementarias de la secuencia trasera (3’UTR) del ARNm, sin afectar
a la integridad del mismo. Fueron descubiertos por primera vez en el
nematodo Caenorhabditis elegans. Constituyen una subclase de microARN.
Véase Dicer, microRNA, trailer sequence y translational
repression.
snoRNA : ARNnop.
![]() small
nucleolar RNA.
small
nucleolar RNA.
snRNA : ARNnp.
![]() small
nuclear RNA.
small
nuclear RNA.
snRNP : RNPnp.
![]() small
nuclear ribonucleoprotein.
small
nuclear ribonucleoprotein.
snurp : snurp.
Voz coloquial derivada del acrónimo inglés snRNP (small
nuclear ribonucleoprotein).
solid-phase peptide sequencing: secuenciación
de polipéptidos en fase sólida.
Método de secuenciación de polipéptidos en el que el polipéptido
cuya secuencia se desea conocer es inmovilizado en una columna especial y degradado,
aminoácido por aminoácido y ciclo tras ciclo, de suerte que en
cada ciclo se detecta, por una parte, el aminoácido resultante de la degradación
y, por otra, el resto de polipéptido que aún no ha sido degradado.
splice site : sitio de corte
y empalme, sitio de empalme, sitio de ayuste.
1 Secuencia de nucleótidos situada a cada extremo de
un intrón. Determina el punto de empalme, es decir, el nucleótido
exacto en el que se producirá la escisión del intrón
y el posterior empalme de exones. Los sitios de empalme se desglosan
a su vez en dos tipos:
a) 5’-splice site, donor splice site, donor site, left splice
site (sitio de empalme 5’, sitio de ayuste 5’; sitio
donador, sitio izquierdo de empalme o ayuste): zona del extremo 5’ del
intrón que contiene la secuencia consenso GU.
b) 3’-splice site, acceptor splice site, acceptor site,
right splice site (sitio de empalme 3’, sitio de ayuste
3’; sitio aceptor, sitio derecho de empalme o ayuste): zona del
extremo 3’ del intrón que contiene la secuencia consenso
AG.
2 Secuencia de nucleótidos que el aparato de empalme
o ayuste reconoce a efectos de la maduración del ARN.
Observación: según la definición 2, se
considera asimismo un sitio de empalme el lugar de ramificación.
Véase branch site, intron y lariat.
spliceosome : empalmosoma,
ayustosoma.
Complejo ribonucleoproteico responsable de la eliminación de los
intrones de los transcritos primarios en el núcleo celular. Consta
de ribonucleoproteínas nucleares pequeñas (RNPnp)
o snurps, formadas a su vez por la
asociación de seis a diez proteínas con moléculas
de ARN pequeñas (ARNnp) ricas en uridinas (se conocen distintos
tipos: U1, U2, U4, U5, U6, U11 y U12). Además de las RNPnp, pueden
formar parte del empalmosoma entre 40 y 100 proteínas o factores
de empalme diversos. Véase intron.
Observación: a partir del momento en que la traducción
más difundida de splicing es «corte y empalme» o «empalme» a
secas (y, más recientemente, «ayuste»), lo lógico
es que este complejo ribonu-cleoproteico se llame como se indica y
no «espliceosoma», como se observa en algunos libros de
texto.
splicing : corte y empalme;
escisión y empalme; empalme; ayuste.
1 RNA splicing (corte y empalme de ARN): en las
células eucariotas, es el proceso postranscripcional, autocatalítico
o enzimático de eliminación de las secuencias no codificantes
o intrones y de reunión de las secuencias codificantes o exones
del:
a) ARN nuclear heterogéneo (ARNnh) para formar el ARN mensajero
continuo (ARNm) que se ha de traducir en proteína;
b) ARN ribosómico (ARNr);
c) ARN de transferencia (ARNt).
También se ha descrito el fenómeno de empalme o ayuste
en los ARNt de procariotas y bacteriófagos. Véase intron.
2 Protein splicing (empalme de proteínas):
modificación postraduccional de una proteína precursora.
Conlleva dos escisiones proteolíticas concertadas y un ligamiento,
que redunda en la eliminación de una secuencia interna de la
cadena polipeptídica original (inteína) para formar una
proteína madura. Se cree que es un proceso autocatalítico.
3 DNA splicing (empalme de ADN) o gene splicing (empalme
de genes): la unión covalente de dos fragmentos de ADN bicatenario.
Desde el punto se vista enzimático, se trata de un ligamiento
de dos fragmentos de ADN catalizado por una ADN-ligasa.
Observaciones: la traducción más popular al español
de la expresión RNA splicing es «corte y empalme»,
pese a que la voz splicing significa literalmente «empalme» o «acoplamiento».
En este caso, a veces, el verbo to splice se utiliza con partículas
como out o in para referirse a la eliminación o desempalme de
intrones (spliced out) o al empalme de exones (spliced
in, spliced
together) propiamente dichos; el término inglés splicing,
no obstante, encierra ambos significados, de supresión de intrones
y de reunión de exones, a la vez. Hay registro de su traducción
por «empalme» o «ayuste» a secas, entendiéndose
por ello el empalme de los exones de ARN. «Ayuste», una
voz de origen náutico que significa «costura y unión
de dos cabos», ya figura en ciertos libros de biología
molecular como una traducción posible de splicing.
splicing junction : zona
de unión, sitio de unión.
![]() splice
site.
splice
site.
splicing site : sitio de
corte y empalme, sitio de empalme, sitio de ayuste.
![]() splice
site.
splice
site.
SRP: SRP.
![]() signal
recognition particle.
signal
recognition particle.
SSB protein: proteína
SSB.
![]() single stranded DNA binding
protein
single stranded DNA binding
protein
ssDNA : ADNmc.
![]() single-stranded
DNA.
single-stranded
DNA.
ssRNA : ARNmc.
![]() single-stranded
RNA.
single-stranded
RNA.
stable ternary complex: complejo
ternario estable.
En la transcripción, es la asociación de ARN, ADN y ARN-polimerasa
que ha dejado atrás el promotor del gen e ingresa en la fase de
elongación.
startpoint: inicio transcripcional.
![]() transcription start point
transcription start point
STR: STR.
![]() short
tandem repeats.
short
tandem repeats.
strand : cadena, hebra.
1 Ordenación lineal de nucleótidos unidos por
enlaces fosfodiéster.
2 Ordenación lineal de aminoácidos unidos por
enlaces peptídicos.
start codon : codón
de iniciación.
Triplete que marca el inicio de un marco de lectura abierto y, por ende,
el comienzo del mensaje contenido en el gen. Casi siempre es «AUG» (el
triplete que codifica la metionina), pero en los organismos procariotas
también puede ser «GUG».
Sting domain : dominio Sting.
![]() Piwi
box.
Piwi
box.
stop codon : codón
de terminación, codón de finalización de lectura,
codón de parada.
Codones reconocidos por un factor de terminación de la traducción
debido a que carecen de un anticodón complementario. Cuando el
factor los reconoce, se interrumpe la traducción y se libera el
polipéptido nuevo. En el código genético universal,
son los codones «UAG», «UGA» y «UAA».
stRNA: ARNtp.
![]() small
temporal RNA.
small
temporal RNA.
stRNA precursor: precursor
del ARNtp.
![]() short
hairpin RNA.
short
hairpin RNA.
structural gene: gen estructural.
Cualquier gen que codifica una proteína o un ARN. Los productos
de estos genes desempeñan una gran diversidad de funciones, por
ejemplo, pueden ser proteínas estructurales, enzimas o incluso proteínas
o ARN con función reguladora.
STS: STS.
![]() sequence-tagged
sites.
sequence-tagged
sites.
substrate: sustrato.
1. Especie química cuya reacción con otro reactivo
químico se observa.
2. Molécula o entidad química cuya conversión
en un producto o en una serie de productos es catalizada por una o
varias enzimas. También se conoce como «reactante» (reactant)
de la reacción química.
3. Solución o mezcla en polvo de todos los ingredientes
o elementos necesarios para el crecimiento de un cultivo de microorganismos
o de la formación de un producto.
4. Componente de un medio nutritivo que proporciona
los elementos necesarios para el crecimiento de un microorganismo
(p.ej.: carbono, nitrógeno, etc.).
sugar-phosphate backbone: esqueleto
de azúcares y fosfatos.
![]() backbone.
backbone.
super-integron : superintegrón.
![]() integron.
integron.
supression: supresión.
![]() supressor
mutation.
supressor
mutation.
supressor mutation: mutación
supresora.
Mutación que compensa otra mutación con la consiguiente
restauración casi total o parcial del fenotipo normal en el
doble mutante. Se distinguen dos categorías: intergénicas
(intergenic supressor mutations) e intragénicas (intragenic
supressor mutations). Las intergénicas están localizadas
en un gen distinto del gen en el que se produjo la mutación
primaria (tal sería el caso si, por ejemplo, ocurriera una mutación
en el anticodón del ARNt correspondiente a un codón mutado
del ARNm, de modo que ahora el ARNt es capaz de leer ese codón
e insertar un aminoácido de función equivalente en la
proteína que confiere el fenotipo). Las intragénicas
están localizadas en el mismo gen en el que se produjo la mutación
primaria (tal sería el caso de las mutaciones que restauran
el marco de lectura original o producen una sustitución de aminoácido
en un sitio distinto de donde se produjo la primera mutación,
de suerte que compensa la desaparición del primero). La mutación
supresora se diferencia de la retromutación propiamente dicha
en que no restituye completamente la función génica primitiva,
sino que en los dobles mutantes sólo ocurre una reversión
parcial del fenotipo primitivo. Véanse reverse
mutation y reversion.
symport: cotransporte unidireccional.
Traslado de dos solutos de un lado a otro de una membrana biológica
de forma simultánea y en la misma dirección. Véase
cotransport.
Observación: en los libros de texto se traduce con frecuencia
por «simporte», pero en esos casos casi siempre se especifica
que es un cotransporte unidireccional.
symporter: simportador.
Transportador de dos solutos en idéntica dirección. Véase
porter.
synonymous codons : codones
sinónimos.
Codones que codifican el mismo aminoácido, aunque difieren en
su secuencia de nucleótidos. Son codones sinónimos, por
ejemplo, UUU y UUC, pues ambos codifican la fenilalanina.