paramutation: paramutación.
Silenciamiento de la expresión de un alelo activo por parte de
otro inactivo situado en el mismo locus en ciertos heterocigotos. Los
alelos que inducen el silenciamiento se llaman «paramutágenos» (paramutagenic),
el alelo sensible al silenciamiento se llama «paramutable» (paramutable)
y las formas alteradas del alelo paramutable se denominan «paramutadas» o «paramutantes» (paramutant).
La paramutación produce en un aumento progresivo de metilaciones
de citosinas en el alelo paramutable. Se trata de un fenómeno
de naturaleza epigenética, estable y heredable (pues los alelos
no se reactivan durante la meiosis o la mitosis), descubierto en el maíz.
Véase homology-dependent gene silencing
(HDGS) y locus.
paratope: parátopo.
Región del anticuerpo que se une con el epítopo de un antígeno.
Tiene una forma complementaria de la del epítopo antigénico específico,
de modo que este último encaja a la perfección y ello facilita
la formación de múltiples enlaces no covalentes con el parátopo.
Así, varios aminoácidos de las regiones hipervariables de las cadenas
pesadas y ligeras del anticuerpo establecen contacto con el antígeno.
Véase ANTIBODY.
passenger DNA: ADN clonado.
![]() cloned
DNA.
cloned
DNA.
PAZ domain: dominio PAZ.
Dominio de unos 110 aminoácidos que toma su nombre de las tres
familias de proteínas en las que se ha encontrado: Piwi, Argonauta
y Zwille/Pinhead. También es común a ciertas proteínas
de la diferenciación celular y de la ribointerferencia tales como
CAF, Sting y Dícer.
PCR: PCR.
![]() polymerase
chain reaction.
polymerase
chain reaction.
pentose-phosphate backbone : esqueleto
de pentosas y fosfatos.
![]() backbone.
backbone.
peptide backbone : esqueleto
peptídico.
![]() backbone.
backbone.
peptide bond : enlace peptídico.
Enlace covalente resultante de una reacción de condensación
entre el grupo carboxilo a de un aminoácido y el grupo amino a
de otro, con pérdida de una molécula de agua. Los aminoácidos
de una proteína se enlazan entre sí mediante enlaces peptídicos.
peptide linkage : enlace
peptídico.
![]() peptide
bond.
peptide
bond.
permease: permeasa.
Proteína o grupo de proteínas que facilita el traslado
de un soluto (biomoléculas, iones o proteínas) de un lado
a otro de una membrana biológica mediante un mecanismo de transporte
activo (con gasto de energía).
Observación: pertenecen a la clase 2 (transferasas) o
3 (hidrolasas) de la clasificación de la IUBMB. La oligopéptido-permeasa,
por ejemplo, cataliza la siguiente reacción:
ATP + H2O + oligopéptidoexterior =
ADP + fosfato+ oligopéptidointerior.
El nombre común de esta última enzima es oligopeptide-transporting
ATPase (ATPasa transportadora de oligopéptidos) y pertenece
a la subcategoría EC 3.6.3.23 de la IUBMB.
phage: fago.
Abreviatura de bacteriófago. Véase bacteriophage.
phagemid: fagómido.
Vector de clonación mixto, con características de un plásmido
y de un fago filamentoso (por lo general, M13 o f1, pero también
puede contener secuencias derivadas del fago λ).
Recibe asimismo el nombre de fásmido (phasmid). Contiene
sitios de inicio de la replicación del plásmido y del fago:
en el interior de una célula hospedadora funciona como un plásmido
normal, que se replica y cuyas copias se reparten en el momento de la
división celular de forma controlada entre las células
hijas, pero si la célula que lo alberga es infectada por un fago
filamentoso adecuado (el fago auxiliar o helper), el fagómido
cambia su modo de replicación como resultado de la presencia del
producto del gen II del fago auxiliar en la célula; esta proteína
reconoce la secuencia ori(+) de la región intergénica del
fagómido, produce una muesca en el ADNbc e inicia la replicación
de éste por el modelo del círculo rodante, igual y de forma
simultánea que el ADNbc del fago auxiliar. De esta manera, la
célula acaba exportando dos tipos de viriones, de aspecto idéntico
pero distinto contenido, que contienen el ADNmc genómico del fago
auxiliar o una de las hebras del fagómido. Los viriones que portan
el fagómido son infecciosos y pueden inyectar el vector en células
susceptibles, donde se vuelven a comportar como plásmidos bacterianos
normales. El fagómido se utiliza para sintetizar sondas monocatenarias,
secuenciar un fragmento clonado y en experimentos de mutagénesis
dirigida. Su principal atractivo consiste en la posibilidad de clonar
fragmentos de ADN monocatenario largos, con poco riesgo de pérdida
de segmentos por deleción. Son ejemplos de fagómidos los
vectores pUC118 y pUC119, λZAP y los de la serie pBluescript.
phasmid: fásmido.
![]() phagemid
phagemid
phenotype: fenotipo.
1 Conjunto de características funcionales y estructurales
de un individuo que resultan de la interacción de su genotipo
con el medio ambiente. Véase genotype.
2 Características codificadas por los alelos de uno o
varios locus en estudio.
phosphodiester backbone : esqueleto
de enlaces fosfodiéster.
![]() backbone.
backbone.
phosphodiester bond: enlace
fosfodiéster.
Unión establecida mediante dos enlaces éster entre una
molécula de ácido fosfórico (H3PO4)
y dos radicales R’ y R’’. Estos radicales son grupos químicos
que contienen carbonos. En los enlaces fosfodiéster de las hebras
de los ácidos nucleicos participan el carbono de la posición
5’ de la pentosa (ribosa o desoxirribosa) y el carbono de la posición
3’ de la pentosa contigua. Estos enlaces son asimismo característicos
de los glicerofosfolípidos.
physical map: mapa
físico.
Asignación de un lugar específico en el genoma a determinadas
secuencias conocidas de nucleótidos, de modo que sirvan de puntos
de referencia (marcas) para la futura secuenciación genómica.
Estas secuencias no deben estar demasiado separadas ni demasiado esparcidas,
y deben conservar una distancia entre sí que pueda conocerse con
razonable precisión. En la actualidad existen varios métodos
para construir mapas físicos de los genomas, a saber, clone
mapping, radiation hybrid mapping, fluorescent in situ
hybridisation (FISH), long-range restriction mapping, sequence-tagged
site (STS) mapping y expressed sequence tags (EST) mapping
Piwi box : dominio Piwi.
Dominio conservado de unos 40 a 80 aminoácidos descubierto por
primera vez en el extremo carboxilo de las proteínas Piwi —forma
abreviada de P-element induced wimpy testis— y Sting de Drosophila.
Forma parte de un dominio estructural más grande (de 300 aminoácidos),
también muy conservado, que está presente, incluso, en
los genomas procariotas. Se desconoce su estructura y función,
pero suele caracterizar a las proteínas que participan en la ribointerferencia
y el mantenimiento de las células precursoras de la línea
germinal de Drosophila.
plantibody: fitoanticuerpo.
Anticuerpo de origen humano o mamífero sintetizado por una planta
transgénica.
plasmid: plásmido.
Molécula de ADN bicatenario circular que se multiplica de forma
independiente del ADN cromosómico de su hospedador natural, la
célula bacteriana (y más raramente otros microorganismos).
Tiene un tamaño variable entre unas 1-5 kb y más de 100
kb y es portador de genes de resistencia a antibióticos, producción
de toxinas y enzimas. Se replica en la célula hospedadora antes
de la división celular y por lo menos una de las copias se transmite
a cada célula hija, pero pueden existir de 1 a 50 copias o más
por célula. No se consideran plásmidos ni el ADN mitocondrial
ni el ADN de los cloroplastos. Se utilizan con suma frecuencia como vectores
de clonación en ingeniería genética; en estos casos
se reduce su tamaño natural al mínimo a fin de poder insertarles
el ADN que se desea clonar y mejorar la frecuencia de obtención
de recombinantes (la transformación con plásmidos de tamaño
superior a 15 kb es extremadamente ineficaz). Entre los vectores plasmídicos
más típicos y populares figuran los de la serie pBR (especialmente
el pBR322), derivada del plásmido ColE1 de E.coli, que
sintetiza a la colicina E1.
plasmid vector: vector plasmídico.
Plásmido transformado en vector de clonación. Véase
plasmid.
-plast: plasto.
Sufijo derivado del
griego que entra en la composición de diversos términos
botánicos de origen griego. Designa una célula (como en bioplasto o protoplasto),
un corpúsculo organizado o una partícula organizada (como en cloroplasto,
amiloplasto) o bien se relaciona con formar o plasmar. En botánica
se utiliza mucho en función sustantiva como sinónimo estricto de
plastidio. Véase PLASTID.
plastid: plastidio, plástido.
Cualquier
miembro de una familia de orgánulos presentes únicamente
en el citoplasma de las células vegetales, que desempeñan una función
de reserva, de fotosíntesis o de biosíntesis de moléculas
esenciales para el funcionamiento celular. Contienen ADN y ribosomas, están
delimitados por una membrana doble y pueden sintetizar algunas proteínas
propias. Cada plastidio deriva de un precursor común, el proplastidio
(proplastid), presente en el meristema vegetal. El proplastidio se diferencia
en un tipo específico y, en determinadas condiciones, cada tipo específico
es capaz de desdiferenciarse, así como de interconvertirse en otros tipos
plastidiales. Los plastidios varían sobremanera en número, tamaño,
forma, contenido y función según el tipo celular y el estadio de
desarrollo de la célula, y se reproducen por fisión, con independencia
del ciclo celular. Son ejemplos de plastidios los cloroplastos (contienen clorofila),
los aleuroplastos (contienen aleurona), los amiloplastos (acumulan gránulos
de almidón), los cromoplastos (contienen pigmentos de color, como los
carotenos y las xantofilas), los eleoplastos (contienen lípidos) y los
leucoplastos (plastidios incoloros implicados en la síntesis de monoterpenos;
no deben confundirse con los amiloplastos). Véase -PLAST.
plastidome: plastidoma.
Conjunto de
plastidios de una célula vegetal. Véase PLASTOME.
plastome: plastoma.
Genoma de un plastidio.
Observación: en botánica, el término plastoma se
utiliza a veces como sinónimo de plastidoma. En cambio, en biología
molecular, la palabra plastoma se usa preferentemente para referirse al
genoma de un plastidio. Véase PLASTIDOME.
plus strand : cadena positiva.
![]() coding
strand.
coding
strand.
Observación: la denominación «cadena positiva» (plus
strand) y «cadena negativa» (minus strand) se
debe reservar para designar las cadenas codificante y no codificante
de los genomas víricos, respectivamente.
point mutation: mutación
puntual.
Cambio de una base por otra en un triplete de nucleótidos (codón)
que deriva en la codificación de un aminoácido distinto
(missense mutation) o en la creación de un codón
de terminación prematura de la síntesis de una proteína
(nonsense codon). Véanse nonsense
codon, nonsense mutation y missense
mutation.
poly(A) : poli(A).
Abreviatura de «poliadenilato».
poly-A signal: señal
de poliadenilación.
Secuencia de nucleótidos de los genes eucariotas codificadores de
proteínas que, una vez transcrita, desencadena la unión de
una serie de factores al ARNm precursor a efectos de la escisión
del futuro ARNm del ARNm precursor y de la poliadenilación del extremo
5’ del ARNm recién escindido por parte de la polinucleótido-adenilil-transferasa
o poli(A)-polimerasa (PAP).
Observación: mientras esto sucede, la ARN-polimerasa sigue
elongando la molécula de ARNm precursor restante (que puede alcanzar
varios centenares y hasta miles de nucleótidos de largo) antes de
disociarse del ADN poniendo fin a la transcripción. Esta segunda
molécula de ARN se degrada en el núcleo de inmediato.
poly(A)
tail : cola de poli(A).
Serie de adenilatos añadidos en el extremo 3’ de los ARNm
eucarióticos (excepto los ARNm de las histonas) y de algunas bacterias.
La enzima que cataliza la adición se denomina «polinucleótido-adenilil-transferasa» o «poli(A)-polimerasa».
polyadenilation : poliadenilación.
Adición de una secuencia de poliadenilatos en el extremo 3’ de
un ARN eucariótico que acaba de ser traducido.
polycistronic mRNA : ARNm
policistrónico.
Molécula de ARNm que determina la síntesis de varios productos
génicos debido a que contiene más de un marco de lectura
abierto. Es característico de los organismos procariotas.
polymerase : polimerasa.
Nombre común con el que se designan las enzimas que forman polímeros
de nucleótidos.
polymerase chain reaction: reacción
en cadena de la polimerasa.
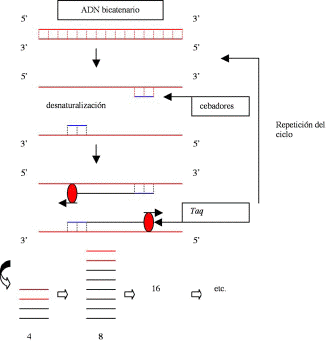 Método
para sintetizar grandes cantidades de un segmento específico
de ADN a partir cantidades inferiores a 1 µg del ADN de muestra
(y en teoría a partir de una sola molécula de ADN). La
PCR multiplica («amplifica») exponencialmente una secuencia
específica de ADN bicatenario. Comprende varios ciclos divididos
en tres etapas (desnaturalización, hibridación y extensión
del cebador) que se resumen de la siguiente manera: primero se desnaturaliza el
ADN por calor (aprox. a 90 ºC), y luego se deja bajar la
temperatura de modo que los extremos 3’ de las hebras ahora separadas
se puedan aparear con oligodesoxinucleótidos perfectamente complementarios,
cuya longitud sea suficiente (20-30 nucleótidos) para formar
híbridos estables con la molécula que sirve de plantilla.
Estos oligodesoxinucleótidos funcionan como cebadores (primers),
de modo que una polimerasa resistente a la desnaturalización
por calor, la polimerasa Taq —llamada así por la bacteria
termófila Thermus aquaticus, de la que se había
aislado—, extiende los extremos 3’ de ambos oligonucleótidos
utilizando las hebras del ADN bicatenario como plantilla, proceso que
se conoce como «extensión del cebador» (primer
extension). Una última desnaturalización pone fin
a un ciclo y da comienzo al siguiente. Al cabo del primer ciclo se
obtienen dos moléculas bicatenarias del ácido nucleico
original. El ciclo anterior se repite muchas veces (el número óptimo
es de 20 a 50 veces en una PCR típica), sin añadir más
enzima y usando las moléculas obtenidas en el ciclo anterior
como plantilla. De esta forma se produce un aumento exponencial del
número de copias de la secuencia específica igual a 2n,
donde n es el número de ciclos. Varios factores son críticos
para la PCR: la concentración de nucleótidos, la especificidad
y la longitud de los cebadores (entre 18 y 30 bases) y la concentración
del ión magnesio. Por sus características intrínsecas,
el método es extremadamente sensible a la presencia de moléculas
de ADN contaminante, que puede dar lugar a amplificaciones inespecíficas
(aparición de «falsos positivos»).
Método
para sintetizar grandes cantidades de un segmento específico
de ADN a partir cantidades inferiores a 1 µg del ADN de muestra
(y en teoría a partir de una sola molécula de ADN). La
PCR multiplica («amplifica») exponencialmente una secuencia
específica de ADN bicatenario. Comprende varios ciclos divididos
en tres etapas (desnaturalización, hibridación y extensión
del cebador) que se resumen de la siguiente manera: primero se desnaturaliza el
ADN por calor (aprox. a 90 ºC), y luego se deja bajar la
temperatura de modo que los extremos 3’ de las hebras ahora separadas
se puedan aparear con oligodesoxinucleótidos perfectamente complementarios,
cuya longitud sea suficiente (20-30 nucleótidos) para formar
híbridos estables con la molécula que sirve de plantilla.
Estos oligodesoxinucleótidos funcionan como cebadores (primers),
de modo que una polimerasa resistente a la desnaturalización
por calor, la polimerasa Taq —llamada así por la bacteria
termófila Thermus aquaticus, de la que se había
aislado—, extiende los extremos 3’ de ambos oligonucleótidos
utilizando las hebras del ADN bicatenario como plantilla, proceso que
se conoce como «extensión del cebador» (primer
extension). Una última desnaturalización pone fin
a un ciclo y da comienzo al siguiente. Al cabo del primer ciclo se
obtienen dos moléculas bicatenarias del ácido nucleico
original. El ciclo anterior se repite muchas veces (el número óptimo
es de 20 a 50 veces en una PCR típica), sin añadir más
enzima y usando las moléculas obtenidas en el ciclo anterior
como plantilla. De esta forma se produce un aumento exponencial del
número de copias de la secuencia específica igual a 2n,
donde n es el número de ciclos. Varios factores son críticos
para la PCR: la concentración de nucleótidos, la especificidad
y la longitud de los cebadores (entre 18 y 30 bases) y la concentración
del ión magnesio. Por sus características intrínsecas,
el método es extremadamente sensible a la presencia de moléculas
de ADN contaminante, que puede dar lugar a amplificaciones inespecíficas
(aparición de «falsos positivos»).
Observación: la PCR es
una de las técnicas más utilizadas de la ingeniería
genética en la actualidad. Produce un resultado similar al de
la clonación del ADN, dado que multiplica («amplifica»)
de forma selectiva un fragmento de ácido desoxirribonucleico.
Debemos este invento a Kary Mullis, quien, como él mismo relata
en su autobiografía, tuvo la genial idea cuando conducía
de regreso a casa con un amigo en abril de 1983, época en que
buscaba una modificación del procedimiento de secuenciación
de Sanger-Coulson («didesoxi» o enzimático). Mullis
dio a conocer la PCR en una reunión científica en 1984,
y en 1993 recibió el Premio Nobel de Química por este
descubrimiento.
polynucleotide: polinucleótido.
Polímero de nucleótidos unidos por enlaces 5’-3’ fosfodiéster
de longitud superior a 50 unidades. Los de menor longitud se denominan oligonucleótidos.
Véase oligonucleotide.
porter: transportador.
Proteína o grupo de proteínas de membrana que facilita
el traslado de un soluto de un lado a otro de una membrana biológica,
con o sin gasto de energía. Se diferencia en tres tipos básicos,
según el número de solutos transportados y su dirección
de traslado: si transporta dos solutos distintos de forma simultánea
(o secuencial) y en direcciones opuestas se llama «antiportador» (antiporter),
cuando transporta dos solutos distintos de forma simultánea (o
secuencial) y en la misma dirección se denomina «simportador» (symporter),
y si solamente transporta un sustrato a la vez recibe el nombre de «uniportador» (uniporter).
Observación: en algunos diccionarios especializados de
lengua inglesa (el Singleton, entre otros), porter figura como
sinónimo no sólo de transporter, sino también
de permease, aunque generalmente este último término
se reserva casi siempre para los sistemas de transporte activo (con
gasto de energía). Véase permease.
positive regulator: activador.
![]() activator
activator
post-transcriptional
gene silencing (PTGS) : silenciamiento génico postranscripcional.
Degradación citoplasmática del ARNm de un gen específico
debido a la presencia de ARNbc complementarios a él. Puede acompañarse
de metilaciones en el gen específico. Son fenómenos de
silenciamiento génico postranscripcional la cosupresión,
la extinción (quelling) y la ribointerferencia. Véase co-suppression, Homology-dependent gene silencing
(HDGS), quelling y
RNA interference.
PPD proteins : proteínas
PPD.
![]() Argonaute
proteins.
Argonaute
proteins.
Observación: el acrónimo PPD proviene del nombre «PAZ
and Piwi Domain». Véase PAZ domain y Piwi
box.
pre-mRNA : preARNm.
![]() precursor
mRNA.
precursor
mRNA.
pre-RISC : preRISC.
Complejo RISC antes de su activación con ATP. Véase RNA-induced
silencing complex y RNA interference.
pre-RNA : preARN.
![]() precursor
RNA.
precursor
RNA.
pre-stRNA : preARNtp.
![]() short
hairpin RNA.
short
hairpin RNA.
precursor mRNA (pre-mRNA) : ARNm
precursor (preARNm).
Molécula de ARN procedente de la transcripción inmediata
de un gen y cuyo producto funcional será una proteína.
Con este nombre se designa a veces el ARN nuclear heterogéneo
(ARNnh) y otras veces, sobre todo en los organismos eucariotas, cualquiera
de las formas intermedias de un ARN primario en vías de convertirse
en un ARN mensajero maduro (ARNm). Véase heterogeneous
nuclear RNA.
precursor RNA : ARN precursor.
Molécula de ARN procedente de la transcripción inmediata
de un gen y cuyo producto funcional puede ser tanto una proteína
como un ARN. Véase primary transcript.
presequence : presecuencia.
![]() leader
sequence.
leader
sequence.
Pribnow box: caja de Pribnow.
Secuencia de seis nucleótidos TATAAT extremadamente conservada de
los promotores procariotas reconocida por el factor σ (sigma) de iniciación
de la transcripción, que se sitúa a unas 10 bases de distancia
del extremo 5’ del nucleótido que marca el inicio de la transcripción.
Es una secuencia consenso y recibe este nombre en honor a uno de sus descubridores,
David Pribnow, pues con frecuencia se ignora que fueron dos (David Pribnow
y Heinz Schaller). Su equivalente en los genes eucariotas es la caja de
Hogness. Véanse consensus sequence, Hogness
box y TATA box.
primary
transcript : transcrito
primario.
Molécula de ARN procedente de la transcripción inmediata
de un gen y cuyo producto funcional puede ser tanto una proteína
como un ARN. En algunos organismos procariotas, los transcritos primarios
son también transcritos maduros cuando no experimentan modificaciones
tras su transcripción (ARNm, ARNr o ARNt). En los organismos eucariotas,
no obstante, todos los transcritos primarios sufren algún tipo
de modificación, por lo que nunca son iguales a los transcritos
maduros. En este último caso, las expresiones «transcrito
primario» y «ARN precursor» son sinónimas. Véase
precursor RNA.
primase: primasa.
ARN-polimerasa especializada en la replicación del ADN. Cataliza
la formación de novo de pequeños fragmentos de ARN
(los cebadores) sobre una de las hebras de ADN que sirve de plantilla.
Se activa al asociarse con otras proteínas que participan en la
replicación del ADN, como la ADN-helicasa. Véase DNA
replication, primer y RNA polymerase.
Observación: esta enzima debería pertenecer en teoría
a la clase EC 2.7.7.6 (ARN-polimerasas dirigidas por ADN) de la nomenclatura
enzimática del Comité de Nomenclatura de la Unión
Internacional de Bioquímica y Biología Molecular (NC-IUBMB),
sin embargo no está recogida como tal ni en ésa ni en ninguna
otra clase de dicha nomenclatura. Por otro lado, aunque desde el punto
de vista enzimático pueda ser equivalente a otras ARN-polimerasas
celulares, conviene mantener su identidad como una ARN-polimerasa distinta
dada su función peculiar en la replicación del ADN, que
no es intercambiable por la de ninguna otra ARN-polimerasa.
primer: cebador.
Oligonucleótido de 5 a 10 nucleótidos de longitud cuyo
3’-OH
utiliza la ADN-polimerasa dirigida por ADN como punto de partida para
la síntesis de ADN.
primosome: primosoma.
Complejo de proteínas indispensable para la actividad de
la enzima primasa. Posibilita la síntesis de los fragmentos de
Okazaki sobre la cadena retrasada del ADN que se está replicando.
probe : sonda.
Cualquier fragmento de ADN o ARN que ha sido marcado con radionúclidos,
fluoróforos u otras moléculas, como la biotina o la digoxigenina,
con objeto de detectar secuencias complementarias en experimentos de
hibridación molecular. Se obtiene por polimerización in
vitro a partir de una secuencia complementaria o por purificación
de un fragmento de restricción de un ácido nucleico natural
o clonado. Salvo indicación contraria, son fragmentos de ADN.
Cuando el fragmento es de ARN recibe el nombre de «ribosonda» (riboprobe).
Observación: no es lo mismo que ‘grupo indicador’.
Véase reporter group.
processive enzyme: enzima
procesiva, enzima asociativa.
Enzima o complejo enzimático que sintetiza o degrada de forma progresiva
un biopolímero y realiza varios ciclos de la misma reacción
catalítica sin disociarse del sustrato (o de la plantilla o de ambos
a la vez) tras cada reacción catalítica. Véanse processive
enzyme y processivity.
Observación: en ciertos libros de texto de bioquímica
traducidos al castellano figura indistintamente como «enzima progresiva» y «enzima
procesiva». Una ADN-polimerasa procesiva típica es capaz de
añadir un promedio de 1000 nucleótidos por segundo al hidroxilo
del extremo 3’ del cebador.
processivity: procesividad,
capacidad de procesamiento.
Capacidad de una enzima o del complejo enzimático de llevar a
cabo múltiples ciclos catalíticos de forma progresiva sin
disociarse de su sustrato polimérico (en vez de disociarse tras
cada reacción catalítica). Es una medida de la eficacia
de la enzima.
Observación: es una de las propiedades de las enzimas cuyos
sustratos son de naturaleza polimérica. En el caso de la ADN-polimerasa,
la procesividad de la enzima (alta, media, baja) se define como el número
de nucleótidos añadidos en promedio cada vez que la enzima
se asocia con el cebador y la hebra plantilla (la procesividad varía
desde unos pocos nucleótidos hasta más de 50 000 nucleótidos
añadidos por complejo de asociación).
proofreading : corrección.
1 En la replicación del ADN, es la actividad exonucleasa
de 3’ a 5’ de una polimerasa, que cataliza la hidrólisis
de uno en uno de los nucleótidos no complementarios de la cadena
plantilla.
2 En la síntesis de proteínas, es la hidrólisis
de un aminoacil-adenilato o de un aminoacil-ARNt por parte de la aminoacil-ARNt-sintetasa
cuando se une un aminoácido equivocado.
3 Asimismo en la síntesis de proteínas, es la
liberación del aminoacil-ARNt del sitio ‘A’ del ribosoma
justo antes de la transposición de este último sobre
el ARNm, cuando el anticodón del ARNt no se ha apareado correctamente
con el codón del ARNm.
prokaryon: procarion.
Núcleo primitivo compuesto de un ADN genómico que no está delimitado
por una membrana de fosfolípidos.
Observación: según Fernando Navarro, en griego, karyon era
palabra llana, de modo que etimológicamente debe escribirse «procarion»,
sin tilde. En la práctica probablemente sea más frecuente
la forma aguda «procarión», quizás por influencia
del francés.
prokaryote: organismo procariota.
Organismo unicelular cuyas células poseen un núcleo primitivo.
Son organismos procariotas las bacterias (dominio Eubacteria o Bacteria)
y los organismos del dominio Archaea en la clasificación
de los seres vivos en tres dominios, o lo que es lo mismo, todos los
organismos del reino Monera, en la clasificación de los
seres vivos en cinco reinos. Véase prokaryon.
Observación: el uso valida asímismo las denominaciones «organismo
procarionte» y «organismo procariótico», aunque
de los tres calificativos sinónimos (procarionte, procariota y procariótico)
el primero esté registrado en el diccionario académico como adjetivo,
el segundo como sustantivo (con remisión al adjetivo «procarionte»,
que es curiosamente donde se define la voz) y el tercero no figure.
(La Academia en este caso no es congruente consigo misma, pues sí recoge
con función adjetiva las voces sinónimas «eucarionte», «eucariota»
y «eucariótico», plegándose al uso.) Una búsqueda en las páginas de
español de Google demuestra que las tres denominaciones gozan de cierta
popularidad, con una ligera preferencia por «organismo procariota»
(organismo procarionte: 14; organismo procariota: 26; organismo procariótico:
14, a 31 de mayo del 2004). En biología molecular es harto frecuente
la sustantivación de estas voces; así, suele hablarse de «los procariotas»
(598 páginas en Google, a 31 de mayo del 2004) o de «los procariontes»
(167 páginas en Google, a 31 de mayo del 2004), pero casi nunca de
«los procarióticos». El DRAE2001 ha dejado constancia de esta costumbre
al menos en la entrada «procarionte», con la abreviatura «U.m.c.s.»
(úsase más como sustantivo). .
prokaryotic: procariótico,
procariota, procarionte.
Relativo a un organismo unicelular que posee un núcleo primitivo.
Véase prokaryote.
promoter: promotor.
Secuencia de ADN bicatenario reconocida por la ARN-polimerasa y otros
factores de transcripción, necesaria para el inicio de la transcripción
del gen contiguo.
Observación: en los organismos procariotas, el promotor
interacciona directamente con la holoenzima con actividad ARN-polimerasa
(unida a una proteína, el factor σ); en los organismos eucariotas,
el promotor contiene secuencias de reconocimiento de factores de
transcripción específicos que, al unirse al promotor
a través de esas secuencias, forman un complejo proteico reconocido
por la ARN-polimerasa, que entonces se fija alrededor del nucleótido
que marca el inicio de la transcripción. En estos organismos,
los promotores reconocidos por las ARN-polimerasas I y II están
localizados en su mayoría antes del nucleótido de inicio
de la transcripción (startpoint, flecha negra en la
figura), pero la mayoría de los promotores de la ARN-polimerasa
III se sitúan después del punto de inicio, esto es,
dentro de la región codificante.
 |
| Una de las posibles configuraciones de un promotor (indispensable para la transcripción de un gen) y un potenciador (dispensable) de un gen transcrito por la ARN-polimerasa II. El promotor contiene varias secuencias dispersas (caja TATA, caja Inr y caja BRE) reconocidas por factores de transcripción. La separación entre el potenciador y el promotor puede ser de varias kb. El potenciador contiene secuencias de reconocimiento de factores de transcripción más juntas. El DNA de la región potenciadora puede plegarse, de modo que los factores estén en contacto directo con los del promotor. El nucleótido del ADN que marca el inicio de la cadena de ARN se denomina «inicio transcripcional» (transcription start site o startpoint) y se designa con el número «+1» (flecha). |
prosthetic group: grupo
prostético.
Cofactor orgánico de una enzima unido a la misma mediante enlaces
fuertes (el ejemplo típico es el grupo hemo). Véase cofactor.
protein backbone : esqueleto
proteico.
![]() backbone.
backbone.
protein intron : inteína.
![]() intein.
intein.
protein splicing : empalme
de proteínas, ayuste de proteínas.
![]() splicing.
splicing.
PTGS : PTGS.
![]() post-transcriptional
gene silencing.
post-transcriptional
gene silencing.