nascent: incipiente, nuevo,
naciente.
Adjetivo que califica a una molécula en vía de síntesis
o que acaba de ser sintetizada.
a) nascent RNA (ARN incipiente): molécula de ARN
en vía de síntesis;
b) nascent RNA (ARN nuevo): molécula de ARN recién
sintetizada;
c) nascent polypeptide (polipéptido naciente):
polipéptido en vía de síntesis que emerge por
el sitio P del ribosoma.
NBF: dominio
de unión a nucleótido.
![]() nucleotide-binding
fold.
nucleotide-binding
fold.
negative regulator: represor.
![]() repressor
repressor
nested genes : genes anidados.
Genes situados dentro de los intrones de otros genes en los genomas eucariotas.
Los genes anidados pueden a su vez contener o no intrones. En este último
caso, posiblemente sean copias retrotranscritas de algún gen.
Constituyen cerca del 6 % del genoma humano.
nested PCR: PCR interna,
PCR anidada.
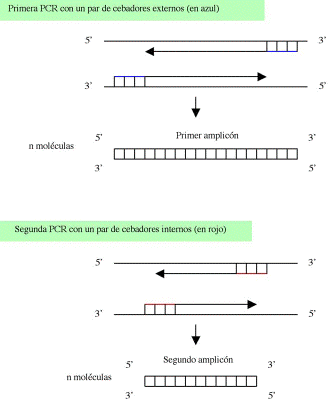 Técnica
que comporta dos reacciones en cadena de la polimerasa (PCR) sucesivas,
con dos pares de cebadores distintos, de tal modo que los cebadores
utilizados en la segunda PCR (internal o nested PCR)
flanqueen una región genómica amplificada en la primera
reacción en cadena (external PCR), como ilustra la figura.
El método de la PCR interna se utiliza sobre todo cuando se
tienen pequeñas cantidades del ADN de interés o cuando
se quieren evitar las amplificaciones inespecíficas que a veces
se observan con la PCR clásica, dado que en cada etapa se realiza
un número menor de ciclos (las PCR de muchos ciclos conllevan
a menudo errores de lectura y de síntesis, debido, entre otras
cosas, a la falta de fidelidad de la polimerasa Taq). Además,
si el producto de la primera PCR es el resultado de una amplificación
inespecífica, el segundo par de cebadores internos no reconocerá la
secuencia complementaria, y por consiguiente no habrá amplificación.
Véase amplicon.
Técnica
que comporta dos reacciones en cadena de la polimerasa (PCR) sucesivas,
con dos pares de cebadores distintos, de tal modo que los cebadores
utilizados en la segunda PCR (internal o nested PCR)
flanqueen una región genómica amplificada en la primera
reacción en cadena (external PCR), como ilustra la figura.
El método de la PCR interna se utiliza sobre todo cuando se
tienen pequeñas cantidades del ADN de interés o cuando
se quieren evitar las amplificaciones inespecíficas que a veces
se observan con la PCR clásica, dado que en cada etapa se realiza
un número menor de ciclos (las PCR de muchos ciclos conllevan
a menudo errores de lectura y de síntesis, debido, entre otras
cosas, a la falta de fidelidad de la polimerasa Taq). Además,
si el producto de la primera PCR es el resultado de una amplificación
inespecífica, el segundo par de cebadores internos no reconocerá la
secuencia complementaria, y por consiguiente no habrá amplificación.
Véase amplicon.
Observación: aunque el método en sí comprende
dos reacciones en cadena de la polimerasa consecutivas, lleva el nombre
de la segunda PCR (la internal PCR o nested PCR) por
ser esta segunda reacción la que aporta el amplicón de
interés. En castellano es más frecuente la denominación «PCR
anidada», donde el adjetivo «anidado» se utiliza
en sentido figurado como sinónimo de «interno»,
en referencia a los cebadores que se hibridan con regiones internas
del primer amplicón.
new genetics: ingeniería
genética.
![]() genetic
engineering.
genetic
engineering.
nitrogen base: base nitrogenada
Molécula orgánica con características básicas
derivada de la purina o de la pirimidina. Si deriva de la purina se denomina
adenina (A) o guanina (G), y si deriva de la pirimidina, timina (T) o citosina
(C). Forman parte de un núcleosido y de los ésteres fosfóricos
de éstos (los nucleótidos) que conforman los ácidos
nucleicos. Véase nucleoside.
NMD: NMD.
![]() nonsense
mediated decay.
nonsense
mediated decay.
noncoding sequence : secuencia
no codificante.
1 En una molécula de ADN, cualquiera de los intrones
o secuencias intergénicas. Véase intron.
2 En el ARN mensajero, es la porción de la secuencia
de nucleótidos que no se ha de traducir en un polipéptido.
Véase utr, 5’utr y 3’utr.
non-coding strand : cadena
no codificante.
![]() noncoding
strand.
noncoding
strand.
noncoding strand : cadena
no codificante, hebra no codificante.
Una de las dos cadenas de ácido nucleico bicatenario (ADNbc, ARNbc)
que sirve de plantilla para la transcripción del ARN. Es sinónimo
estricto de «cadena plantilla» (template strand) en
su tercera acepción.
Observación: la JCBN (Joint
Commission on Biochemical Nomenclature) y la NC-IUB (Nomenclature
Commission of the International Union of Biochemistry and Molecular
Biology) prefieren
esta designación (noncoding strand) a cualquiera de las otras
denominaciones posibles (antisense strand, minus
strand, complementary strand, non-coding strand, template strand, anticoding
strand y transcribing
strand). No obstante, no faltan quienes prefieren utilizar el nombre
de «cadena plantilla» (template strand) por considerar
que no deja lugar a dudas desde el punto de vista conceptual.
nondistributive enzyme: enzima
asociativa.
![]() processive enzyme
processive enzyme
nonprocessive enzyme: enzima
no procesiva, enzima disociativa.
Enzima o complejo enzimático que sintetiza o degrada de forma
progresiva un biopolímero y realiza varios ciclos de la misma
reacción catalítica disociándose del sustrato (o
de la plantilla o de ambos a la vez) tras cada reacción catalítica.
Véanse processive enzyme y processivity.
nonreciprocal translocation: transposición.
![]() translocation.
translocation.
nonrepetitive DNA: ADN no
repetitivo.
ADN formado por secuencias nucleotídicas presentes una sola
vez o en muy pocas copias en el genoma. Cuando se lo desnaturaliza tiende
a volver a renaturalizarse o a reasociarse muy despacio. Es el único
componente de los genomas procariotas y un componente importante de los
genomas eucariotas. Constituyen el 60 % del genoma humano.
nonsense codon : codón
de terminación, codón de finalización de lectura,
codón de parada.
![]() stop
codon.
stop
codon.
Observación: se desaconseja utilizar la expresión «codón
sin sentido» como sinónimo de «codón de terminación»,
pues induce a error, ya que los codones de terminación o parada
cumplen la función precisa de finalizar la traducción
de proteínas (luego, tienen un significado o «sentido»).
nonsense mediated decay (NMD): degradación
mediada por mutaciones terminadoras.
Proceso de eliminación de ARNm portadores de una mutación
terminadora (nonsense mutation) o de una mutación del marco
de lectura (frameshift mutation) que derivan en la creación
de codones de finalización precoz de la síntesis de una
proteína. De no eliminarse estos ARNm se producirían polipéptidos
defectuosos (incompletos) al ser traducidos. Se trata de un fenómeno
asociado a la ribointerferencia o a la HDGS. Véase homology-dependent
gene silencing (HDGS), nonsense mutation, RNA
interference.
nonsense mutation: mutación
terminadora.
Mutación puntual que convierte un codón codificante en
un codón de terminación en el ARNm transcrito. De esta
manera, una mutación puntual en el codón UAU (tyr) que
lo transforme en UAG (codón de terminación, denominado «ámbar» por
motivos históricos) redunda en la interrupción de la síntesis
de una proteína en el lugar donde debería insertarse la
tirosina en el polipéptido original (wild-type). Un cambio
de marco de lectura puede conllevar asimismo la aparición de un
codón de terminación. Véase frameshift
mutation,
nonsense codon, point
mutation y sense codon.
nonsense strand : cadena
no codificante.
![]() noncoding
strand.
noncoding
strand.
non-synonymous codons: codones
no sinónimos.
Dícese de los codones que codifican aminoácidos distintos.
Véase synonymous codons.
non transcribed spacer : espaciador
no transcrito, espaciador intergénico.
Secuencia en el genoma que separa dos unidades de transcripción
y no se transcribe. Suele designar la secuencia separadora de genes en
un agrupamiento.
Observación: este espaciador no se ha de confundir con
un espaciador intragénico (transcribed spacer) ni con
un intrón (intron). Véase transcribed
spacer e intron.
nontranscribing strand : cadena
codificante.
![]() coding
strand.
coding
strand.
nuclease: nucleasa.
Cualquier enzima de la subclase EC 3.1 que hidroliza los enlaces éster
de los ácidos nucleicos. Se clasifica a su vez en endonucleasa
o exonucleasa, según la posición del enlace fosfodiéster
que hidroliza, y en ribonucleasa o desoxirribonucleasa, según
el ácido nucleico que sirva de sustrato. Existen nucleasas que
reconocen específicamente los ácidos nucleicos monocatenarios
o bicatenarios e incluso las hélices híbridas de ADN y
ARN. Véase endonuclease, exonuclease,
deoxyribonuclease y ribonuclease.
nucleic acid : ácido
nucleico.
Polímero de nucleótidos unidos por enlaces 5’-3’ fosfodiéster.
Según que se trate de ribonucleótidos o de desoxirribonucleótidos
se llama ARN o ADN, respectivamente. Desempeña distintas funciones
en las células de los organismos vivos tales como el almacenamiento
de la información genética y su transferencia a la generación
siguiente (ADN) o la expresión de esta información durante
la síntesis de proteínas (ARNm y ARNt), es componente estructural
de orgánulos celulares como los ribosomas (ARNr), cataliza ciertas
reacciones químicas (ribozimas) y participa en mecanismos de regulación
de la expresión génica (mediante ARN complementarios de ARNm
o de ARNbc en la ribointerferencia).
nucleic
probe : sonda nucleica.
![]() probe.
probe.
nucleoside : nucleósido.
Compuesto orgánico constituido por una base nitrogenada (púrica,
derivada de la purina, o pirimidínica, derivada de la pirimidina)
enlazada mediante el nitrógeno 1 de la pirimidina o el nitrógeno
9 de la purina al carbono 1 de una 2-desoxi-D-ribosa o de una D-ribosa
a través de un enlace N-glucosídico de configuración
ß. Según que el azúcar sea la ribosa o la desoxirribosa,
el nucleósido resultante se denomina ribonucleósido (ribonucleoside)
o desoxirribonucleósido (deoxyribonucleoside). Los nucleósidos
más comunes de los sistemas biológicos son la adenosina,
la guanosina, la citidina y la uridina (que contienen una ribosa) y la
dexosiadenosina, la desoxiguanosina, la desoxicitidina y la timidina (que
contienen una dexosirribosa).
nucleosome: nucleosoma.
Subunidad estructural de la cromatina. Consta de un octámero de
histonas y de un segmento de ADN de aproximadamente 200 pares de bases
que se enrolla sobre el octámero dando casi dos vueltas alrededor
de él, como un hilo a su carrete. El octámero está compuesto
a su vez de dos moléculas de cada una de las histonas H2A, H2B,
H3 y H4. Véase chromatin, core DNA, core
particle, histone y linker
DNA.
nucleotide : nucleótido.
Éster fosfórico de un nucleósido. Consta de uno o
más grupos fosforilo que esterifican el grupo hidroxilo en las posiciones
3’ o 5’ (con mayor frecuencia) del azúcar correspondiente
(ribosa o desoxirribosa). Los fosforilos se clasifican en grupos fosfatos
α, ß o γ con arreglo a su proximidad al azúcar. Según que
el azúcar sea la ribosa o la desoxirribosa, el nucleótido
resultante se denomina ribonucleótido (ribonucleotide) o
desoxirribonucleótido (deoxyribonucleotide). La base nitrogenada
es la portadora de la información genética, mientras que
los grupos fosfato y los azúcares desempeñan una función
estructural. Los ribonucleótidos de las células de los organismos
son el ácido adenílico, el ácido guanílico,
el ácido uridílico, el ácido citidílico, y
los desoxirribonucleótidos el ácido desoxirriboadenílico,
el ácido desoxirriboguanílico, el ácido desoxirribocitidílico
y el ácido timidílico.
Observación: con suma frecuencia, en los textos de bioquímica,
tanto en inglés como en castellano, los desoxirribonucleótidos
se escriben prescindiendo de la partícula –ribo, así es
frecuente ver escrito: desoxiadenílico, desoxiguanílico
y desoxicitidílico. Véase nucleoside.
nucleotide-binding fold: dominio
de unión a nucleótido, dominio de unión a ATP.
Observación: el nucleótido que habitualmente se
une a este dominio de unión es la ATP, por este motivo generalmente
se considera sinónimo de ATP-binding domain (dominio
de unión a ATP). Véase ATP-binding
domain.