idiotope: idiótopo.
Epítopo o determinante antigénico
situado en las regiones variables de las cadenas livianas y pesadas (VH y VL,
respectivamente) de un anticuerpo (en la zona de contacto con el antígeno o alrededor de
esta zona). Es sinónimo de determinante idiotípico. Véanse ANTIBODY e IDIOTYPE.
idiotype: idiotipo.
Conjunto de idiótopos
presentes en las regiones variables de las cadenas livianas y pesadas (VH y VL,
respectivamente) de un anticuerpo. Pueden estimular la producción de anticuerpos antiidiotípicos.
Véanse ANTIBODY e IDIOTOPE.
idiotypic determinant: determinante
idiotípico.
→ IDIOTOPE
immunogen: inmunógeno.
Antígeno capaz de suscitar una respuesta inmunitaria adaptativa (humoral
o celular) al ingresar en un organismo inmunocompetente. No todos los antígenos
son inmunógenos. Véase ANTIGEN.
immunoglobulin: inmunoglobulina.
→ ANTIBODY
inducer: inductor.
Molécula que induce la síntesis de las enzimas responsables
de su metabolismo.
initiation codon : codón
de iniciación.
![]() start
codon.
start
codon.
initiator tRNA : ARNt iniciador.
Metionil-ARNt que reconoce específicamente el codón de
inicio de la traducción de una proteína —generalmente
AUG, pero en las bacterias también puede ser GUG o UUG— en
el sitio P del ribosoma. Pese a tener el anticodón UAC específico
de la metionina, no puede reconocer los codones AUG del interior del
ARNm, porque su estructura se lo impide. En los organismos procariotas,
la metionina unida a este ARNt está formilada y el ARNt iniciador
se indica con el símbolo ARNtfMet (tRNAfMet);
en los organismos eucariotas, en cambio, la metionina no está formilada
y el ARNt iniciador se suele indicar con el símbolo ARNtiMet (tRNAiMet).
Tanto en los eucariontes como en los procariontes, el símbolo
del ARNt que reconoce los AUG internos es ARNtmMet (tRNAmMet).
(Estas convenciones de escritura pueden presentar ligeras variantes.).
insert: inserto.
Fragmento de ADN heterólogo insertado en un vector de clonación.
Suele ser sinónimo de «ADN clonado». Véase cloned
DNA.
insertion: inserción.
Introducción de uno o más pares de bases en una molécula
de ácido nucleico. Es un tipo de mutación que suelen producir
los colorantes de acridina o los transposones.
insertion sequence: secuencia
de inserción.
![]() transposable
element.
transposable
element.
insulator : aislador
de la cromatina.
Secuencia de ADN de los organismos eucariotas que impide la diseminación
del efecto activador o inactivador de la transcripción de un gen
al demarcar el límite del radio de acción de un potenciador
sobre el gen o bien impedir la transformación de la cromatina en
heterocromatina cerca del gen que debe permanecer activo. Véase
enhancer.
Observación: también se conoce con los nombres de boundary
element y chromatin boundary.
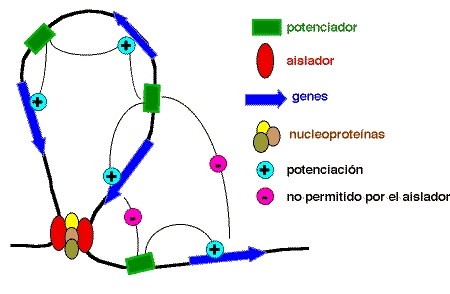 |
| Representación esquemática de la interferencia de un aislador. |
integron: integrón.
Elemento de los genomas bacterianos con capacidad de capturar y expresar
genes exógenos que confieren ventajas selectivas a la bacteria
hospedadora (muchos de estos genes exógenos confieren resistencia
a antibióticos). Puede ser móvil (transposónico
o plasmídico) o fijo (cromosómico). En su forma más
sencilla consta de tres componentes necesarios para la captura y la
expresión del gen exógeno (también denominado «casete
génico»): un gen codificante de la integrasa intI (que
es una recombinasa), un lugar de recombinación específico attI para
la integración del gen exógeno y por lo menos un promotor Pant para
la expresión de ese gen. En esta configuración, y sin
casete génico, tiene un tamaño aproximado de 1,1 kb.
Observación: descubiertos a principios de 1980, los integrones
son elementos antiguos que han ido evolucionando a la par que el genoma
bacteriano. Hubo quien los definió como «sistemas de expresión
y adquisición de genes» (gene acquisition and expression
systems), siendo el casete génico la unidad de adquisición
o captura de ADN. En la actualidad, se clasifican en nueve clases según
la secuencia de la integrasa componente: las clases In1 a In3 contienen
casetes génicos de resistencia a antibióticos; las clases
In4 a In7 contienen casetes que no codifican resistencia a antibióticos,
la clase In8 carece de casete génico y la clase In9 contiene un
casete de resistencia a antibióticos y otros casetes génicos
de función desconocida. Pueden contener múltiples casetes
génicos incorporados en tándem (se denominan entonces «superintegrones»;
puede haber hasta 150 casetes en tándem en los superintegrones
cromosómicos, cada uno flanqueado por sitios de recombinación 59-be),
además de genes de resistencia que no son «casetes» (es
decir, no son móviles, sino que son componentes permanentes o
fijos del integrón). La inserción o escisión de
casetes génicos de resistencia en un integrón dado desempeña
una función importante en la incorporación y formación
de nuevas recombinaciones de genes de resistencia a los antibióticos.
El hecho de que muchos integrones posean más de un casete génico
de resistencia y de que algunos integrones se localicen a su vez dentro
de elementos móviles o transferibles, como son los transposones
(p.ej.: Tn21 o Tn1696) y los plásmidos, que
también pueden contener genes de resistencia a antibióticos,
hace que la selección de uno de los genes de resistencia del integrón
conlleve la selección de los demás genes de resistencia
(fenómeno conocido como «selección en autoestop» de
los genes ligados). Se han descubierto integrones en numerosas especies
de bacterias, incluidas las de la familia Enterobacteriaceae y
otras bacterias gramnegativas, como Vibrio cholerae y Pseudomonas
aeruginosa, o grampositivas, como Corynebacterium glutamicum,
etc.
intein : inteína.
Secuencia interna de aminoácidos que se elimina de una proteína
precursora recién traducida por medio de una reacción de
transpeptidación. Véase splicing y transpeptidation.
Observación: este nombre deriva por apócope y
aféresis de la expresión intervening protein sequence y
equivale conceptualmente a los intrones de los ácidos nucleicos.
intergenic DNA : ADN
intergénico.
ADN de los genomas eucariotas que separa los genes entre sí. Lo
conforman secuencias de diversas clases, en ocasiones extremadamente
repetidas, como sucede en los genomas de las plantas. El ADN intergénico
constituye un gran porcentaje del genoma de numerosos organismos, incluido
el de los seres humanos, y no carece necesariamente de función.
Algunos autores consideran que los promotores forman parte del ADN intergénico
(en este caso, el ADN intragénico constaría solamente de
exones e intrones). Véase junk DNA.
intergenic supressor mutation: mutación
supresora intergénica.
![]() supressor
mutation.
supressor
mutation.
intervening sequence : secuencia
intercalada, secuencia interpuesta.
![]() intron.
intron.
intragenic supressor mutation: mutación
supresora intragénica.
![]() supressor
mutation.
supressor
mutation.
intrinsic terminator: terminador
intrínseco, terminador independiente de ρ (ro).
![]() Rho-independent
terminator.
Rho-independent
terminator.
intron
: intrón.
1 Secuencia intragénica de desoxirribonucleótidos
que se transcribe pero no se conserva en la molécula de ARN
madura (ARNm, ARNr o ARNt).
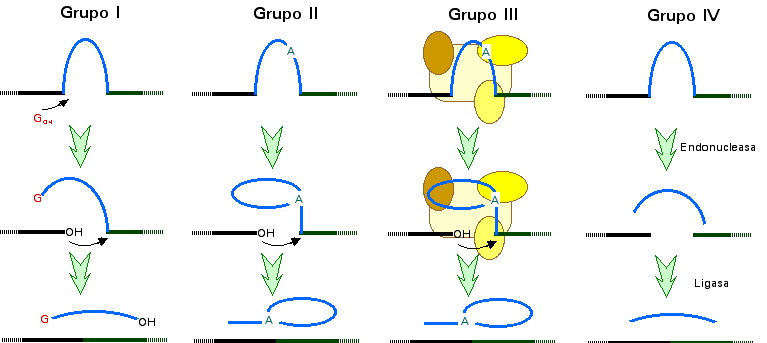
2 En el transcrito primario, es la secuencia de ribonucleótidos
que se elimina durante el proceso de corte y empalme (ayuste), y que,
por lo tanto, no forma parte del transcrito maduro (ARNm, ARNr, ARNt).
Se distinguen cuatro tipos según su forma de eliminación
durante el proceso de corte y empalme (ayuste) y la clase de genes en
los que se observan. Los intrones de los grupos I, II y III se escinden
mediante reacciones de transesterificación. Los intrones del grupo
I (presentes en los genes de ARNr de algunos organismos eucariotas inferiores –como,
por ejemplo, Tetrahymena thermophila–, en los genes mitocondriales
de hongos y en ciertos fagos) se caracterizan por carecer de secuencias
consenso en los sitios de empalme, aunque pueden tenerlas en su interior,
y eliminarse por un proceso autocatalítico estrechamente relacionado
con la estructura secundaria y terciaria de la molécula precursora
de ARN (en esta clase de intrones, una guanosina o un nucleótido
de guanosina libre –guanilato, GMP, GDP o GTP– aporta el grupo
OH que produce la primera transesterificación; véase la
figura siguiente); los intrones del grupo II (presentes en genes mitocondriales
de hongos) disponen de sitios de empalme con secuencias consenso (responden
a la regla GT-AT) y, al igual que los del grupo I, se eliminan mediante
un proceso autocatalítico dependiente de la estructura secundaria
y terciaria del ARN (en este caso, una adenina cede el grupo 2’OH
que produce la primera transesterificación); los intrones de los
genes nucleares o del grupo III son idénticos a los del grupo
II (responden a la regla GT-AT), pero, a diferencia de éstos,
necesitan de la presencia del empalmosoma (o ayustosoma) para escindirse
(véanse los círculos amarillos y marrones en la figura
de abajo); tanto en el grupo II como en el grupo III se forma una estructura
en lazo característica (lariat) cuando se empalman los
exones; los intrones del grupo IV, presentes en los tRNA eucariotas,
son los únicos que no se eliminan por medio de una reacción
de transesterificación, sino a través de un corte endonucleásico
con ligamiento ulterior. Véanse lariat,
spliceosome y splicing.
Observación: intron es una voz formada por apócope
y aféresis a partir de intervening sequence region. En
los organismos eucariotas superiores, la mayoría de los genes
se hallan interrumpidos por intrones de longitud por lo general mayor
que la de los exones correspondientes. Puede haber intrones en los
genes nucleares que codifican proteínas, en los genes nucleolares
de ARNr o en los genes de ARNt. En los organismos procariotas, los
genes suelen ser continuos, pero se han descubierto intrones en fagos
y en algunos ARNt bacterianos (Véase la ilustración).
 inversion: inversión.
inversion: inversión.
Giro de 180º de un determinado segmento cromosómico, con
el resultado de que la secuencia de un gen en el segmento en cuestión
queda invertida con respecto a la del resto del cromosoma. Estas inversiones
pueden incluir o no el propio centrómero (círculo blanco
en el medio de los segmentos coloreados de la figura de abajo).
isoaccepting tRNAs : ARNt
isoaceptores.
![]() cognate
tRNAs.
cognate
tRNAs.