deletion: deleción.
Pérdida de uno o más pares de bases en una molécula
de ácido nucleico.
Observación: no debe escribirse con doble ce. Esta voz
nos viene del inglés a través del latín deletio. Vertida
al castellano da «deleción» y no «delección».
denaturation : desnaturalización.
Desplegamiento total o parcial de la conformación nativa de un
polipéptido, una proteína o un ácido nucleico. Las
proteínas con estructura terciaria, como lo son casi todas las
enzimas y proteínas que desempeñan funciones de regulación,
se desnaturalizan o despliegan al ser calentadas o cuando varía
el pH de la disolución en la que se encuentran. Puede ser un proceso
irreversible, que se acompaña de la pérdida de la actividad
biológica y de la solubilidad de la molécula. En el caso
de los ácidos nucleicos, no se considera desnaturalización
la pérdida de superenrollamiento, pero sí la desaparición
de los puentes de hidrógeno entre cadenas complementarias.
denature, to : desnaturalizar.
Perder un biopolímero (por ejemplo, una proteína o un ácido
nucleico) su estructura original.
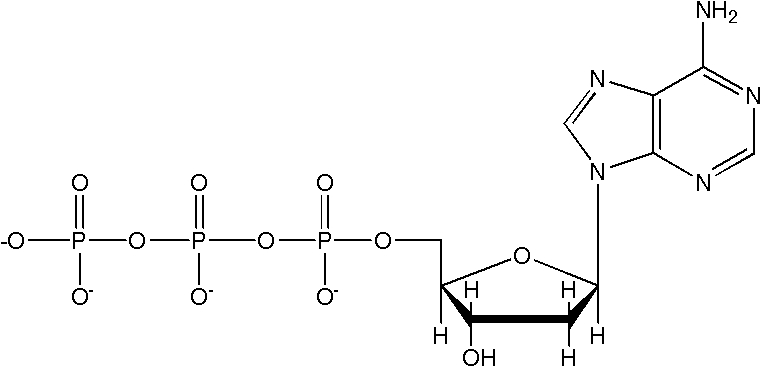
deoxynucleoside triphosphate: desoxinucleósido
trifosfato.
Éster trifosfórico de un nucleósido cuyo azúcar
es la desoxirribosa. Los más comunes son cuatro: la desoxiadenosina-5’-trifosfato
(dATP), la desoxiguanosina-5’-trifosfato (dGTP), la desoxicitidina-5’-trifosfato
(dCTP) y la timidina-5’-trifosfato (dTTP). Véase nucleotide.
 |
| Estructura del nucleótido desoxiadenosina-5’-trifosfato (dATP) |
deoxyribonuclease: desoxirribonucleasa.
Nucleasa que hidroliza los enlaces fosfodiéster del ADN. Véase
endonuclease y exonuclease.
deoxyribonucleic
acid (DNA) : ácido desoxirribonucleico (ADN).
Polímero de desoxirribonucleótidos unidos por enlaces 5’-3’ fosfodiéster.
Es uno de los dos ácidos nucleicos naturales conocidos y la molécula
que almacena la información genética por excelencia en
los seres vivos.
Observación: la estructura tridimensional del ADN es
la de dos largas hebras o cadenas que adoptan en conjunto el aspecto
de una doble hélice antiparalela. Existen asimismo ADN monocatenarios,
tricatenarios y circulares. Véase double
helix y ribonucleic
acid.
deoxyribonucleoside triphosphate: desoxirribonucleósido
trifosfato.
![]() deoxynucleoside triphosphate
deoxynucleoside triphosphate
ddNTP: ddNTP.
→ DIDEOXYNUCLEOSIDE TRIPHOSPHATE
Dicer : Dícer.
Enzima que interviene en los procesos de ribointerferencia (RNA interference)
y de represión de la traducción (translational repression).
Consta de varios dominios, uno con actividad helicasa del ARN, dependiente
de ATP (en el extremo amino), un dominio PAZ, dos dominios contiguos
con actividad endorribonucleasa III (ARNasa III) en serie y un dominio
de unión a ARNbc (en el extremo carboxilo). Desde el punto de
vista evolutivo es una enzima muy conservada. Actúa sobre moléculas
de ácido ribonucleico de dos tipos:
a) ARNbc de 100 o más pares de bases. En este caso, la enzima
divide el ARNbc en fragmentos regulares de 21 a 25 pares de bases conforme
se va desplazando a lo largo de la molécula; este proceso requiere
energía (ATP). Los fragmentos resultantes se denominan «ARN
interferentes pequeños» (small interfering RNA) y
son indispensables para la degradación de ARNm invasores o aberrantes
en el fenómeno de la ribointerferencia. Véase RNA
interference y small interfering RNA.
 b) ARN en
forma de horquilla de aproximadamente 70 nucleótidos (ARNhc).
b) ARN en
forma de horquilla de aproximadamente 70 nucleótidos (ARNhc).
En este segundo caso, la enzima escinde la horquilla y las zonas no
apareadas del ARN horquillado, y libera un fragmento monocatenario
de 21 a 23 nucleótidos
(línea negra). Este fragmento se denomina «ARN temporal
pequeño» (small temporal RNA) y es indispensable
para la regulación postranscripcional de algunos ARNm endógenos.
Véase microRNA, short hairpin RNA, small
temporal RNA y translational
repression.
dideoxy analogue: didesoxirribonucleósido
trifosfato.
→ DIDEOXYNUCLEOSIDE TRIPHOSPHATE.
Observación: en el lenguaje
específico, este análogo
de desoxirribonucleósido fosfato también se conoce más abreviadamente
con el nombre de ‘didesoxianálogo’.
dideoxynucleoside triphosphate: didesoxirribonucleósido
trifosfato.
Cualquier nucleósido trifosfato artificial en el que el grupo hidroxilo
situado en la posición 3’ de la desoxirribosa ha sido sustituido
por un átomo de hidrógeno (se trata, pues, de un 2’,3’-didesoxirribonucleósido-5’-trifosfato).
Al carecer del grupo hidroxilo en la posición 3’ (3’-OH),
no puede formar un enlace fosfodiéster con otro nucleótido en esa
posición y, por consiguiente, la elongación de una cadena de ADN
se interrumpe de inmediato en el lugar donde ha ingresado un 2’,3’-didesoxirribonucleósido
fosfato. El método de secuenciación enzimática ideado por
Sanger se basa en esta propiedad. Véase ENZYMATIC SEQUENCING METHOD.
distributive enzyme: enzima
disociativa.
![]() nonprocessive
enzyme
nonprocessive
enzyme
DNA : ADN.
![]() deoxyribonucleic
acid.
deoxyribonucleic
acid.
DNA backbone : esqueleto
del ADN.
![]() backbone.
backbone.
DNA cloning: clonación
de ADN.
![]() cloning.
cloning.
Observación: con frecuencia se utiliza como sinónimo
de «ingeniería genética». Véase genetic
engineering.
DNA-dependent RNA polymerase : ARN
polimerasa dependiente de ADN.
![]() DNA-directed
RNA polymerase.
DNA-directed
RNA polymerase.
Observación: es una antigua y frecuente denominación
de la enzima cuyo nombre sistemático y recomendado es «ARN
polimerasa dirigida por ADN».
DNA-directed RNA polymerase : ARN
polimerasa dirigida por ADN.
![]() RNA
polymerase.
RNA
polymerase.
DNA fingerprinting: huella
genética.
Tipificación genética de un individuo sobre la base de
variaciones presentes en su secuencia de ADN. En la práctica es
la pauta de distribución de fragmentos de restricción del
ADN en una placa autorradiográfica que, a modo de un código
de barras, es propia de cada individuo.
Observación: en el ámbito de la medicina legal
se conoce más comúnmente con el nombre de DNA profiling,
aunque no sean exactamente lo mismo, pues la última es una huella
genética de segunda generación. Debemos este invento
a Alec J. Jeffreys y cols., que fueron los primeros en concebirla como
prueba de identificación parental o individual en 1985 (individual-specific ‘fingerprints’ of
human DNA). Se basa en el hecho de que en el genoma humano existen
regiones de gran variabilidad que difieren en cada persona (salvo entre
gemelos). Cada una de esas regiones (en color anaranjado, figura a),
también conocidas como «minisatélites de ADN»,
consiste en un número variable de repeticiones de nucleótidos
en tándem (VNTRs, variable number of tandem repeats).
Cada repetición tiene a su vez una secuencia básica en
común (core sequence) con otras VNTR polimórficas,
además de secuencias que le son específicas (véase
la figura 1). Después de purificar el ADN y cortarlo con enzimas
de restricción que dejan intactos los minisatélites,
los fragmentos polimórficos obtenidos se separan por electroforesis
(figura b) y se hibridan con una sonda radiactiva complementaria, ya
sea de las regiones comunes (multi-locus
probe, MLP) o bien de las regiones específicas de las VNTR
(single-locus probe, SLP). Las autorradiografías resultantes
revelan, en el primer caso, una serie de bandas en escalera (cada una
correspondiente a una región hipervariable o a un minisatélite
en particular), y en el último caso, sólo dos bandas,
una que corresponde al alelo paterno y otra al alelo materno del minisatélite
específico. En castellano, la técnica de Jeffreys se
conoce con diversos nombres, a saber: huella digital, impronta de ADN,
huella genómica, identificación dactilar, huella dactilar
del ADN e impronta genética, por citar los más usuales.
Véase DNA profiling, minisatellite y restriction
map.
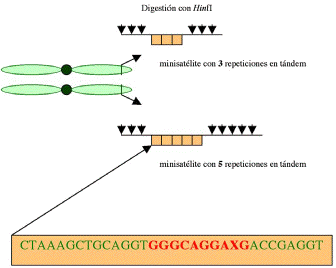 |
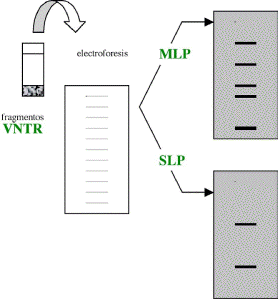 |
| Figura 1. En verde claro se indican dos cromosomas homólogos con minisatélites (VNTR) en sus extremos. El locus correspondiente a la región del minisatélite (banda cromosómica amplificada arriba en color anaranjado) es heterocigoto con respecto a la longitud de la región (un alelo dispone de tres y el otro de cinco repeticiones en tándem). La digestión con HinfI (flechas negras) escinde y separa los minisatélites del resto de la molécula. | Figura 2. Las VNTR (los minisatélites en este caso) procedentes de muchos locus se separan por electroforesis y se transfieren a una membrana de nilón o nitrocelulosa. La hibridación posterior con una sonda que reconoce una secuencia en común de estas VNTR (sonda «multilocus» o MLP) revela una pauta de distribución de fragmentos de ADN semejante a un «código de barras». Si en cambio se utiliza una sonda que reconoce la secuencia específica de un locus dado (sonda «unilocus» o SLP), sólo se observan dos bandas correspondientes al minisatélite específico, una del alelo materno y la otra del paterno. |
DNA ligase: ADN-ligasa.
Enzima que restablece el enlace fosfodiéster roto (muesca) en
una hebra de ADN bicatenario y, a veces, en una hebra de ARN, mediante
la siguiente reacción:
NAD+ + (desoxirribonucleótido)n +
(desoxirribonucleótido)m = AMP + nucleótido
nicotinamídico + (desoxirribonucleótido)n+m
Observación: pertenece a la clase EC 6.5.1.2
del Comité de Nomenclatura de la Unión Internacional de
Bioquímica y Biología Molecular (NC-IUBMB). Su nombre
común es «ADN-ligasa (NAD+)» (DNA ligase [NAD+])
y su nombre sistemático: «poli(desoxirribonucleótido):
poli(desoxirribonucleótido)ligasa (formadora de AMP, formadora
de NMN)» (poly[deoxyribonucleotide]: poly[deoxyribonucleotide]
ligase [AMP-forming, NMN-forming]). También se conoce con
los siguientes nombres:
polydeoxyribonucleotide synthase (NAD), polynucleotide ligase (NAD),
DNA repair enzyme, DNA joinase, DNA ligase (NAD), polynucleotide synthetase
(nicotinamide adenine dinucleotide), deoxyribonucleic-joining enzyme,
deoxyribonucleic ligase, deoxyribonucleic repair enzyme, deoxyribonucleic
joinase, DNA ligase, DNA joinase, deoxyribonucleate ligase, polynucleotide
ligase, deoxyribonucleic acid ligase, polynucleotide synthetase, deoxyribonucleic
acid joinase, DNA-joining enzyme, deoxyribonucleic joinase, deoxyribonucleic
repair enzyme, polynucleotide ligase (nicotinamide adenine dinucleotide),
polydeoxyribonucleotide synthase (NAD+).
DNA polymerase: ADN-polimerasa
Enzima que cataliza la extensión del extremo 3’ de una hebra
de ADN sobre una plantilla de ADN complementario, con liberación
de un pirofosfato (o difosfato, formado por los fosfatos ß y γ del dNTP
recién añadido al extremo 3’-OH de la hebra creciente),
mediante la siguiente reacción:
desoxirribonucleósido trifosfato + DNAn = difosfato
+ DNAn+1
Añade un desoxirribonucleósido trifosfato a la vez y no
puede iniciar la síntesis de ADN de novo, sino que necesita
de un pequeño fragmento de ARN o ADN que sirva de cebador previamente
sintetizado sobre la plantilla, cuyo 3’-OH utiliza para iniciar
la síntesis de ADN. Presenta asimismo actividad exonucleasa en
dirección 3’ a 5’, que cataliza la separación
de los nucleótidos cuyas bases no son estrictamente complementarias
durante la polimerización. Esta actividad de corrección
se conoce en inglés como proofreading.
Observación: pertenece a la clase EC 2.7.7.7 del Comité de
Nomenclatura de la Unión Internacional de Bioquímica y
Biología Molecular (NC-IUBMB). Su nombre común es
«ADN-polimerasa dirigida por ADN» (DNA-directed DNA polymerase)
y su nombre sistemático «desoxinucleósido-trifosfato:
ADN-desoxinucleotidiltransferasa (dirigida por ADN)» (deoxynucleoside-triphosphate:
DNA deoxynucleotidyltransferase [DNA-directed]). También recibe
las siguientes denominaciones: DNA polymerase I, DNA polymerase II,
DNA polymerase III, DNA polymerase α, DNA polymerase ß, DNA polymerase
γ, DNA nucleotidyltransferase (DNA-directed), DNA nucleotidyltransferase
(DNA-directed), deoxyribonucleate nucleotidyltransferase, deoxynucleate
polymerase, deoxyribonucleic acid duplicase, deoxyribonucleic acid polymerase,
deoxyribonucleic duplicase, deoxyribonucleic polymerase, deoxyribonucleic
polymerase I, DNA duplicase, DNA nucleotidyltransferase, DNA polymerase,
DNA replicase, DNA-dependent DNA polymerase, duplicase, Klenow fragment,
sequenase, Taq DNA polymerase, Taq Pol I, Tca DNA polymerase.
DNA polymerase sliding clamp: abrazadera deslizante de
la ADN polimerasa.
Complejo proteico de los organismos eucariotas constituido por diversas
subunidades polipeptídicas que al unirse adoptan la forma de una
rosquilla. Su función es amarrar la subunidad catalítica
de la ADN-polimerasa al ADN conforme se lleva a cabo la replicación
del ADN a gran velocidad y aumentar de este modo la procesividad de la
ADN-polimerasa. Véase processivity.
Observación: la proteína se une firmemente a la
ADN polimerasa en la horquilla de replicación, rodea a la doble
hélice que acaba de sintetizarse (la cavidad central de la proteína
es suficientemente grande para ello), y el complejo formado por la
abrazadera y la ADN polimerasa se desliza a lo largo de la hebra de
ADN conforme avanza la polimerización. Con el amarre de la subunidad
catalítica de la DNA polimerasa a su sustrato, la proteína
impide que la ADN-polimerasa se disocie y abandone el ADN para formar
otro complejo con el cebador y la hebra plantilla, lo cual retardaría
el proceso de síntesis, tal como ocurre en ausencia de la abrazadera.
Una vez que la ADN- polimerasa ha sintetizado el nuevo segmento de
ADN, cambia de conformación y pierde afinidad por la abrazadera
y el sustrato, de modo que se libera del complejo y está lista
para iniciar otro ciclo de polimerización a partir de un nuevo
cebador. En E. coli, un dímero formado por dos subunidades ß de
la ADN-polimerasa III desempeña una función semejante
a la de la abrazadera deslizante de los organismos eucariotas. En el
banco de proteínas Swiss-Prot/TrEMBL el nombre común
de esta proteína es DNA polymerase sliding clamp y no sliding
DNA clamp como figura de forma abreviada en algunos libros de texto
en idioma inglés. En los organismos eucariotas también
recibe el nombre de PCNA (proliferating cell nuclear antigen).
DNA profiling: perfil
de ADN.
Método para identificar individuos por las características únicas
de su ADN. Es, en esencia, una huella genética de segunda generación,
que incorpora una reacción en cadena de la polimerasa (PCR). Se
utiliza mucho en las pruebas de paternidad o para determinar la posible
implicación en un crimen de un sujeto sospechoso. Véase DNA fingerprinting.
Observación: se diferencia de una huella genética
convencional en que no hace falta disponer de una gran cantidad de
ADN en buen estado y en que la muestra (usualmente de sangre o saliva,
o de la mucosa bucal) es objeto de una reacción en cadena de
la polimerasa con cebadores fluorescentes que amplifican determinadas
regiones hipervariables del ADN. Estas regiones, que no están
circunscritas a los extremos de los cromosomas, como en el caso de
los minisatélites, sino que se encuentran dispersas en el genoma,
reciben el nombre de short tandem repeats (STR) o «microsatélites»;
en general, la amplificación de tres o cuatro de estas regiones
(locus) suele ser suficiente para obtener resultados concluyentes.
Luego, el ADN amplificado se separa por electroforesis en un tubo capilar,
y a medida que los fragmentos migran por el capilar un detector lee
las marcas fluorescentes con la ayuda de una fuente de luz láser.
Las señales digitales del láser son a su vez leídas
e interpretadas por un programa informático específico,
que elabora un gráfico. En éste, cada región de
STR se visualiza como dos picos, correspondientes a los alelos paterno
y materno, pero si no hay polimorfismo en esa región de STR
(es decir, si los alelos materno y paterno son de igual longitud),
sólo se visualiza un pico. Véase DNA
fingerprinting, genetic polymorphism, microsatellite y short
tandem repeats.
DNA replication: replicación
de ADN.
Proceso de copia de una molécula de ADN en dos moléculas
idénticas durante la fase S del ciclo celular.
Observación: para que la replicación del ADN tenga
lugar es necesaria la presencia de los cuatro dexosirribonucleósidos
trifosfato –dCTP, dGTP, dATP y dTTP– , una hebra de ADN que
sirva de plantilla, ARN cebadores y varias proteínas con sus
correspondientes cofactores, a saber: ADN-helicasas, proteínas
de unión a ADN monocatenario (proteínas SBB), topoisomerasas,
primasas, ADN-polimerasas, proteínas deslizantes de sujeción
de la ADN polimerasa, ribonucleasas H y ADN ligasas. En los organismos
eucariotas, la replicación del ADN consta sucintamente de las
siguientes etapas:
DNA sequencing with chain-terminating inhibitors: secuenciación
enzimática.
→ ENZYMATIC SEQUENCING METHOD
DNA splicing : corte y empalme
de ADN, ayuste de ADN.
![]() splicing.
splicing.
DNase: desoxirribonucleasa,
ADNasa.
![]() deoxyribonuclease.
deoxyribonuclease.
Observación: curiosamente en inglés se da preferencia
a la grafía DNase por sobre DNAse o DNAase.
dNTP: desoxinucleósido
trifosfato.
![]() deoxynucleoside
triphosphate
deoxynucleoside
triphosphate
domain: dominio.
1. Secuencia continua de aminoácidos de una proteína,
que se dobla o pliega varias veces sobre sí misma hasta formar
una unidad globular o compacta. Se estima que cada dominio puede funcionar
como una unidad o un módulo estable en solución si se
llega a escindir la cadena polipeptídica que los separa. Este
tipo de dominio se denomina dominio estructural.Las proteínas
de tamaño superior a los 20 000 Da constan de dos o más
dominios de este tipo; por ejemplo, la cadena liviana (ligera) de un
anticuerpo tiene dos dominios estructurales.
2. Cualquier región de una proteína asociada
a una función específica, con independencia de su organización
estructural (p.ej., el dominio de unión a un receptor, a un
sustrato –dominio catalítico–, a la membrana celular –dominio
transmembranario–, etc.). Este tipo de dominio se denomina dominio
funcional. Cada dominio funcional puede contener a su vez uno o
más dominios estructurales.
3. Zona o región de la membrana plasmática formada
por una determinada clase de componente (por ejemplo, fosfolípidos).
4. Fragmento, área o región de ADN de tamaño
concreto en el genoma de un organismo (p.ej.: «... the Arabidopsis
Adh gene and the GRF4 gene are contained on discrete domains in the
genome», «... figure 4C illustrates that this gene
resides on a 100-kb domain—a domain clearly distinct from the
fragment occupied by Adh1.»).
donor site : sitio donador.
![]() splicing
site.
splicing
site.
donor splice site : sitio
donador.
![]() splicing
site.
splicing
site.
double helix : doble hélice.
Estructura tridimensional que adoptan las dos hebras del modelo de ADN
propuesto por James Dewey Watson y Francis Harry Compton Crick en 1953
(por el que obtuvieron el Premio Nobel en 1962, junto con Maurice Hugh
Frederick Wilkins). Es prácticamente idéntica a la estructura
del ADN-B: los desoxirribonucleótidos de una hebra se concatenan
mediante la unión del hidroxilo 3’ de una desoxirribosa
con el hidroxilo 5’ de la desoxirribosa adyacente por un enlace
fosfodiéster. Cada desoxirribonucleótido está formado
a su vez por una base nitrogenada, que es la parte variable del ADN
(adenina, citosina, timina o guanina), un azúcar (la desoxirribosa)
y un grupo fosfato. Las dos hebras se mantienen unidas por puentes
de hidrógeno entre bases complementarias (adenina-timina, citosina-guanosina).
En la secuencia de bases reside la información genética.
double-stranded : bicatenario,
de cadena doble, de hebra doble.
Adjetivo que califica a un ácido nucleico formado por dos cadenas
de nucleótidos. Véase double-stranded
DNA, double-stranded
RNA y duplex.
double-stranded DNA (dsDNA) : ADN
bicatenario.
Molécula de ADN en la que dos cadenas de desoxirribonucleótidos
con orientación opuesta (antiparalela) se unen mediante puentes
de hidrógeno entre las bases nitrogenadas. Véase DNA.
Observación: la sigla española, ADNbc, apenas
se utiliza.
double-stranded RNA (dsRNA) : ARN
bicatenario.
1 Molécula de ARN en la que dos cadenas de ribonucleótidos
con orientación opuesta (antiparalela) se unen mediante puentes
de hidrógeno entre las bases nitrogenadas. Constituye el material
genético de algunos virus (por ejemplo, los Rotavirus).
2 Cualquier secuencia de nucleótidos interna en una molécula
de ARN monocatenario que se pliega sobre sí misma y forma apareamientos
entre bases complementarias.
Observación: la sigla española, ARNbc, apenas
se utiliza.
double-stranded RNA interference : ribointerferencia,
interferencia por ARN (iARN).
![]() RNA
interference.
RNA
interference.
dsDNA : ADNbc.
![]() double-stranded
DNA.
double-stranded
DNA.
dsRNA : ARNbc.
![]() double-stranded
RNA.
double-stranded
RNA.
dsRNA-induced gene silencing : ribointerferencia,
interferencia por ARN (iARN).
![]() RNA
interference.
RNA
interference.
dsRNA trigger : ARNbc desencadenante.
![]() trigger, RNA
interference.
trigger, RNA
interference.
duplex : bicatenario, híbrido.
Adjetivo que se aplica a los ácidos nucleicos de dos cadenas o
hebras. Según la clase de nucleótidos que componen estas
cadenas (ribonucleótidos, desoxirribonucleótidos) admite
distintas traducciones:
a) duplex RNA-DNA (ADN-ARN híbrido, doble hélice
híbrida) cuando el ácido nucleico está formado por
una hebra de ADN y otra de ARN;
b) duplex DNA (ADN bicatenario) cuando el ácido
nucleico está formado por dos hebras de ADN (véase double-stranded
DNA);
c) duplex RNA (ARN bicatenario) cuando el ácido
nucleico está formado por dos hebras de ARN (véase double-stranded
RNA).
Observación: los libros de texto también recogen
las variantes «doble» y «dúplex». No
son incorrectas desde el punto de vista semántico, pero sí mucho
menos frecuentes que el adjetivo «bicatenario».
duplex DNA : ADN bicatenario.
![]() duplex.
duplex.
duplex RNA : ARN bicatenario.
![]() duplex.
duplex.
duplication: duplicación.
Repetición de una secuencia de bases en una molécula de ácido
nucleico.